Миф о древних ДНК-генеалогических линиях африканцев (часть 1). Древняя днк
Древняя ДНК: Привет из прошлого Статья на конкурс «био/мол/текст»: Генетический материал, выделенный из доисторических артефактов костей, зубов, волосяных фолликулов и других сохранившихся тканей, как правило, не настолько прост, как это кажется многим, однако уже сегодня функционируют несколько десятков лабораторий, которые не боятся трудностей и пытаются заглянуть в далекое (насколько это возможно) прошлое нашей планеты.  Главный спонсор конкурса — дальновидная компания Генотек.Конкурс поддержан ОАО «РВК». Обратите внимание! Эта работа представлена на конкурс научно-популярных статей «био/мол/текст»-2014 в номинации «Биоинформатика и молекулярная эволюция». Спонсором номинации «Биоинформатика» является Институт биоинформатики. Спонсором приза зрительских симпатий выступила фирма Helicon. Свой приз также вручает Фонд поддержки передовых биотехнологий. Как найти археологическую сенсацию? Не секрет, что главное и основное условие при работе с древней ДНК — это превосходная сохранность материала, которая может быть обеспечена комплексом условий. Поэтому на сегодняшний день наибольшее число научных работ по этой тематике основано на образцах, чей возраст не превышает пятидесяти тысяч лет, причем храниться они должны были в идеальных условиях — минимальной температуре и наименьшей влажности (рис. 1). Условная фаланга пальца, обнаруженная археологом в условной пещере, как правило, лишь начало длинного пути к пониманию того, кем же был этот условный человек, и был ли он человеком в научном смысле этого слова. Причем, дабы не упрощать работу археологов, следует отметить, что на поиск условной фаланги может уйти не один десяток лет, в течение которого сотни практикантов будут просеивать тонны осадочной породы в поисках того, что сможет перевернуть современные представления о прошлом. Но мы не будем забегать настолько далеко, ведь после археологической работы с найденной фалангой предстоит плодотворно поработать еще двум группам людей (рис. 2). Палеогенетики: спасение утопающих — дело рук самих утопающих Вслед за археологами в работу включаются молекулярные биологи, которых в дальнейшем для простоты мы будем называть палеогенетиками. Пожалуй, и книги не хватит для того, чтобы описать те «муки», с которыми столкнутся последние. В помощь им — самые разнообразные ими самими (воистину, спасение утопающих — дело рук самих утопающих! — прим. автора) разработанные методики и подходы.  Рисунок 1. Денисова пещера на Алтае. Фото автора.  Рисунок 2. Фаланга пальца неандертальца, найденная в Денисовой пещере в 2010 году [4]. Первое, недолго думая, они «отобрали» у стоматологов их основное орудие труда. На начальной стадии при работе с костями или зубами* биологи активно используют ненавистную с детства стоматологическую бормашину, которой они высверливают ткань, перетирая ее в муку. При этом их основной интерес вызывают некогда пронизанные кровеносными сосудами элементы ткани — сюда с меньшей вероятностью попадают различные бактерии, разрушающие организм после его смерти и, как правило, приводящие к контаминации (загрязнению) образца древней ДНК своими ДНК-фрагментами. Кроме того, туда не попадает ДНК современных людей, например, археологов и антропологов, описывающих находки. Несомненно, загрязнение современной ДНК мешает дальнейшему анализу: во-первых, при амплификации полимераза будет предпочтительнее «работать» с современными бактериальными молекулами ДНК, обходя стороной побитые временем фрагменты древней ДНК, а во-вторых, впоследствии вам придется фильтровать свои данные от всяких ненужных ни вам, ни высокорейтинговым журналам засоров. Кроме «битвы» с контаминацией и борьбы за стерильные лабораторные условия, палеогенетики сталкиваются и с другими серьёзными проблемами. Например, с фрагментацией геномной и митохондриальной ДНК. Причем фрагменты в лучшем случае оказываются около 100 нуклеотидов. Мне встречалось немало людей, утверждавших, что их образцы обладают прекрасной сохранностью, но на деле они говорили о прекрасной сохранности современной ДНК, засорившей их образцы... Следует отметить, что в некоторых случаях археологические останки действительно могут содержать десятки процентов эндогенной ДНК, но зачастую ученые вынуждены работать с долями процентов — плевелами среди куч современного «генетического мусора». Например, в костных останках денисовского человека было обнаружено примерно 70% эндогенной ДНК [3–5], а при анализе митохондриального генома новосвободненского человека этот показатель составил 0,006%, что говорит о диаметрально отличных условиях внешней среды, в которых находился археологический материал. Естественно, 100 нуклеотидов — это ещё не повод «посыпать голову пеплом», ведь таких кусочков может быть тысячи и миллионы. Только следует помнить, что ДНК, как и любая биомолекула, способна вступать в химические реакции с окружающим миром, тут-то и появляются различные модификации нуклеотидов (особенно по краям фрагментов древней ДНК). Наиболее частая постмортальная мутация — дезаминирование цитозинов (C), приводящая к возникновению урацилов (U) в последовательности древней ДНК, которые при проведении ПЦР многократно копируются «бездушным» ферментом ДНК-полимеразой как тимин (Т). Считается, что интенсивность процессов дезаминирования зависит от условий среды, в которой находятся биологические останки, например, от pH и химического состава почв, влажности и температуры. Под влиянием этих факторов фрагменты древней ДНК расплетаются по краям и отдаются во власть химической модификации. Итак, фрагменты древней ДНК, с трудом и валидолом выделенные из условной фаланги условного человека, попадают в пробирку, где над ними продолжают «колдовать» палеогенетики. Эти крохи генетической информации, этот «привет из прошлого» подготавливается для дальнейшего секвенирования (установления первичной последовательности ДНК)*. Для этой цели используют современные приборы — секвенаторы, позволяющие прочитать и обработать громадный массив геномной информации за сравнительно небольшой отрезок времени. Как хорошо уметь считать! После запуска этих машин и получения первичных, «сырых» данных палеогенетики молитвенно складывают руки, ожидая результатов, которые будут получены группой людей, объединенных специальностью биоинформатика [10, 11]. Именно биоинформатики являются последним звеном при работе с древней ДНК, извлеченной из археологического или палеонтологического материала. Между нами, эта их особенность дает им уверенность в своем превосходстве над остальными звеньями научного эксперимента, что часто ведет к торможению многих блестящих научных проектов. Однако с этим приходится мириться, поскольку современная популяционная генетика невозможна без использования биоинформатических методов, и на Западе это десятки людей, связанных одной целью. Обработка данных секвенирования древней ДНК — не самое тривиальное занятие: несмотря на значительные усилия, в данных по-прежнему остается большой процент современной ДНК, а также неидентифицируемого генетического мусора, который нуждается в тщательной очистке, с привлечением математического аппарата. Удивительно, но именно постмортальные модификации нуклеотидных последовательностей древней ДНК и небольшая длина выделенных из биологического материала ДНК-фрагментов служат превосходным маркером при работе с древней ДНК, позволяя отфильтровать весь современный генетический мусор [12] (рис. 3).  Рисунок 3. Тест на контаминацию современным генетическим материалом [12]. Фактически фильтрация данных древней ДНК — наиболее важная часть биоинформатической работы: именно на этой стадии отсекаются все те последовательности, которые впоследствии могут привести к научной ошибке. Далее следуют стандартные этапы по определению митохондриальных гаплогрупп, Y-хромосомных гаплотипов, поиску древних геномных мутаций и даже сборке нового генома (например, используя уже известные геномы современных людей в качестве «каркаса»). Древние перспективы Идеальные случаи, связанные с превосходной сохранностью археологического материала, приводят к тому, что условная фаланга пальца или бедренная кость, найденные где-то в Сибири, становятся прорывом в понимании истории человечества, заставляющим пересмотреть всю его историю или даже опровергнуть казавшуюся незыблемой теорию его происхождения. Такие находки и такие исследования приводят к появлению не только великолепных научных публикаций, но и позволяют зародить надежду на возрождение прошлого. Увы, современные естественнонаучные методы позволяют лишь приподнять занавесь древней истории нашей планеты, но воскресить его вряд ли смогут. Post Scriptum К сожалению, в России подобные исследования находятся в зачаточном состоянии. Только несколько лабораторий пытаются работать с отечественным палеонтологическим и археологическим материалом, который подобно углеводородам уходит за рубеж. В 2014 году сотрудники Центра «Биоинженерия» РАН и НИЦ «Курчатовский институт» впервые в России отсеквенировали последовательность митохондриальной ДНК (и определили её гаплогруппу) у древнего человека, обитавшего на территории Северного Кавказа в эпоху ранней бронзы [12, 13]. Эта пионерская для России работа, возможно, даст толчок отечественной археологической и палеогенетике. - Мамонты, кости и лекарственная устойчивость: новые технологии позволяют изучать эволюцию возбудителей инфекционных заболеваний;
- Это чума;
- Meyer M., Kircher M., Gansauge M.T., Li H., Racimo F., Mallick S., et al. (2012). A high-coverage genome sequence from an archaic Denisovan individual. Science 338, 222–226;;
- Prüfer K., Racimo F., Patterson N., Jay F., Sankararaman S., Sawyer S. et al. (2014). The complete genome sequence of a Neanderthal from the Altai Mountains. Nature 505, 43–49;;
- Fu Q., Li H., Moorjani P., Jay F., Slepchenko S.M., et al. (2014). Genome sequence of a 45,000-year-old modern human from western Siberia. Nature 514, 445–449;;
- Важнейшие методы молекулярной биологии и генной инженерии;
- Огурцы-убийцы, или как встретились Джим Уотсон и Гордон Мур;
- Код жизни: прочесть не значит понять;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Я б в биоинформатики пошёл, пусть меня научат!;
- Вычислительное будущее биологии;
- Nedoluzhko A.V., Boulygina E.S., Sokolov A.S., Tsygankova S.V., Gruzdeva N.M., Rezepkin A.D., Prokhortchouk E.B. (2014). Analysis of the Mitochondrial Genome of a Novosvobodnaya Culture Representative using Next-Generation Sequencing and Its Relation to the Funnel Beaker Culture. Acta Naturae 6, 31–35;;
- Недолужко Артем. (2014). «Путешествие в Денисову пещеру»..
biomolecula.ru Древняя ДНК Википедия ДНК, выделенная из древнеегипетской мумии возрастом 4000 лет ДНК, выделенная из древнеегипетской мумии возрастом 4000 лет Секвенирование древней ДНК — определение нуклеотидной последовательности (секвенирование, от лат. sequentum — последовательность) применительно к молекулам ДНК, извлечённым из древних биологических образцов, таких как палеонтологические и археологические находки, мумифицированные останки, засохшие остатки растений, копролиты. Анализ нуклеотидных последовательностей, полученных секвенированем древней ДНК, позволяют установить филогенетические отношения между видами и проверять гипотезы о связи изменений в окружающей среде и эволюционных изменений популяций, а также предоставляют информацию для калибровки молекулярных часов[1]. При работе с древней ДНК исследователи сталкиваются со множеством проблем, связанных с сохранностью образцов. ДНК может деградировать с течением времени, химически модифицироваться. Микроорганизмы, участвующие в разложении останков, не только нарушают целостность тканей, но и вносят в образец собственную ДНК, тем самым усложняя процесс выделения древней ДНК и биоинформатического анализа полученных данных. Такие методы, как секвенирование нового поколения и обогащение ДНК-библиотек путём гибридизации позволяют заметно увеличить количество получаемых из образцов информации. Проведен анализ ДНК ряда древних животных, в том числе мамонта и пещерного медведя. Анализ ДНК из человеческих останков позволил выделить новую группу древних людей — денисовцев, а также выявить детали происхождения современных этнических групп. Ряд открытий был сделан в результате анализа древней ДНК болезнетворных микроорганизмов: произведен анализ генома чумной палочки из лондонских захоронений XIV века и гриба фитофторы из образцов XIX века. История Исследования древней ДНК начались в 1984 году с секвенирования фрагмента митохондриальной ДНК (мтДНК) вымершей во второй половине XIX квагги[2], подвида бурчелловой зебры. Было обнаружено, что ДНК не только сохраняется на протяжении более полутора веков, но также может быть частично выделена и секвенирована. Вскоре после этого Сванте Паабо секвенировал образцы, полученные из человеческих мумий.[3][4] В этих исследованиях ученый использовал бактериальное клонирование для амплификации фрагментов ДНК. Оказалось, что большая часть ДНК в образцах имеет бактериальное или грибное происхождение, а поддающаяся амплификации эндогенная ДНК состоит в основном из коротких повреждённых фрагментов многокопийных локусов (например, мтДНК) и составляет малую часть исследованной ДНК. Изобретение полимеразной цепной реакции (ПЦР) позволило амплифицировать даже немногочисленные сохранившиеся фрагменты ДНК и дало толчок к развитию этой области, однако также увеличило чувствительность результатов к контаминации[5]. Области применения Исследование древней ДНК имеет важное значение для таких областей науки как генетика, палеозоология, палеоэпидемология, антропология (особенно палеоантропология) и археология. Также эта область стала частью методологии палеогенетики. Анализ древней ДНК может использоваться для оценки эволюционной близости таксономических групп давно вымерших или сильно изменившихся организмов, конретные родственные связи которых выяснить иными способами крайне затруднительно. Особое внимание здесь уделяется областям высокой изменчивости — участкам ДНК, где мутации происходят часто. В частности, это короткие тандемные повторы (STR) и однонуклеотидные полиморфизмы (SNP). Анализ митохондриальной ДНК дает более точную информацию в контексте этого метода, так как митохондриальная ДНК имеет гораздо большее количество копий по сравнению с ядерной ДНК (около 1000 копий митохондриальных ДНК и 2 копии ядерной ДНК на клетку)[6]. Анализ древней ДНК также позволяет устанавливать половую принадлежность скелетных останков представителей видов, женские и мужские особи которых отличаются набором половых хромосом, что особенно важно, когда другие методы, например антропометрия, не могут дать точного ответа[7]. Данный метод находит свое применение и в контексте палеоэпидемологии. Диагностику заболеваний по скелетным останкам и изучение древних пандемий крайне сложно проводить без анализа ДНК возбудителей, сохранившейся в останках больных. Такие исследования стали возможны только в 1990 годах, что позволило прослеживать как эволюцию инфекционных бактериальных штаммов и вирусов, так и их распространение, а также сопоставлять детектируемые по останкам симптомы болезни с современными знаниями о конкретном заболевании[8][9]. Технические сложности Деградация ДНК Обычно после смерти организма ДНК расщепляется эндогенными нуклеазами. Этого не происходит, если нуклеазы оказываются быстро разрушены или инактивированы, например, вследствие обезвоживания останков, низких температур или большой концентрации соли.[10] Даже в этом случае ДНК со временем повреждается в результате случайного гидролиза или окисления. К гидролитическим повреждениям относятся разрушение фосфатного остова цепи, депуринизация (англ.) (соответствующая позиция остается без азотистого основания) и дезаминирование. Чаще происходит дезаминирование цитозина в урацил, метилированный цитозин (5-метил-цитозин) дезаминируется в тимин; реже аденин превращается в гипоксантин, который комплементарен цитозину, а не тимину, что ведет к неправильному прочтению при секвенировании. Эти повреждения происходят и в живых клетках, однако там они устраняются в процессе репарации. Кроме того, возникают поперечные сшивки между цепями спирали ДНК из-за алкилирования или сшивки ДНК с различными молекулами в результате реакции Майяра. Как и разрывы цепи, поперечные сшивки мешают амплификации при ПЦР[10][5]. Перед секвенированием древнюю ДНК подвергают специальной обработке, чтобы удалить продукты дезаминирования и поперечные сшивки. Средняя длина амплифицируемых фрагментов древней ДНК часто не превосходит 100 пар нуклеотидов, причём для находок из одного места раскопок средняя длина фрагментов убывает с увеличением возраста находки. Современные протоколы секвенирования древней ДНК учитывают эту особенность; в частности, вместо отжига праймеров на фрагменты ДНК к концам фрагментов присоединяются адаптеры, а праймерами служат олигонуклеотиды, комплементарные адаптерам[11]. При низких температурах деградация ДНК идет медленнее, что обеспечивает хорошую сохранность ДНК в образцах, найденных в районе вечной мерзлоты. Однако считается, что даже в идеальных условиях ДНК не может сохраняться на протяжении более 1 млн лет[10][5]. Примесь чужеродной ДНК Часто вследствие загрязнения, или контаминации, лишь малая доля ДНК в образце имеет эндогенное происхождение. Лучшие в этом отношении образцы содержат до 90 % эндогенной ДНК.[1] Археологические образцы всегда содержат некоторое количество ДНК бактерий и грибов, которые колонизируют останки, пока те лежат в земле. Кроме того, в процессе исследования древней ДНК в образец может попасть человеческая или микробная ДНК, присутствующая в любой лаборатории. В отличие от древней ДНК современная хорошо амплифицируется при ПЦР. Продукты ПЦР, содержащие амплифицированную современную ДНК, могут распространиться по лаборатории и увеличить таким образом степень контаминации[1][5]. Для профилактики лабораторной контаминации рекомендуется продолжительная обработка оборудования ультрафиолетом или кислотой и соблюдение требований протокола работы в ПЦР-лабораториях. Исследование древней ДНК не должно проводиться в лаборатории, где ранее изучались современные образцы, особенно близких видов[5]. Многие археологические образцы, особенно найденные до появления современных молекулярно-биологических методов, подверглись контаминации в процессе извлечения и исследования. Если образец был найден несколько десятилетий назад, человеческая ДНК, которой он загрязнен, может обладать всеми признаками древней ДНК. Кости и зубы имеют пористую поверхность, что делает проблематичной их очистку от современной ДНК. Волосы оказываются в этом отношении предпочтительнее: содержащаяся в них ДНК малодоступна для бактерий, а гидрофобная поверхность позволяет очистить их перед извлечением ДНК[1]. В этом случае совпадение последовательностей, полученных в результате независимого исследования разных частей образца (например, бедра и зубов) в разных лабораториях свидетельствует в пользу истинности результата[5]. В случае человеческих останков контаминация современной человеческой ДНК может приводить к ошибкам при исследовании филогении и популяционной генетики. Существует статистический метод, позволяющий отличать древнюю ДНК от современной по паттерну дезаминирования[12][13]. Если известно, что образец ДНК принадлежит женщине, можно картировать отсеквенированные последовательности на Y-хромосому и детектировать таким образом попадание в образец мужской ДНК[14]. Если исследуется ДНК нечеловеческого происхождения, очевидным способом проверки на контаминацию является картирование полученных при секвенировании чтений на человеческий геном, а также на геномы других организмов в случае, если они могли быть источниками контаминации. При секвенировании древней бактериальной ДНК проблема состоит также в том, что известны геномы далеко не всех существующих ныне бактерий, так что из того, что полученная последовательность не входит в известные бактериальные геномы, нельзя заключить, что она принадлежит древней бактерии, а не получена в результате контаминации[5]. Вставки мтДНК и хпДНК в хромосомах Вставки митохондриальной и пластидной ДНК часто встречаются в ядерной ДНК. При исследовании древних образцов ДНК нельзя выделить ДНК органелл, поэтому эти вставки могут быть источником ошибок, так как их не всегда легко отличить по последовательности от настоящей мтДНК или хпДНК. Если такие вставки будут приняты за ДНК органелл, это может исказить результаты исследования филогении или популяционной генетики[5]. Методы обработки древней ДНК В качестве источника древней ДНК часто стараются использовать сохранившиеся части тела, не представляющие большого интереса с точки зрения анатомического строения. Из-за описанных выше процессов в большинстве древних образцов содержание интересующей исследователей древней ДНК очень мало — всего около 1 %. Это количество сильно варьирует в зависимости от природы образца. Последние исследования показывают, что значительно больший выход эндогенной ДНК из останков предков человека достигается при выделении материала из зубов и каменистой части височной кости.[15] Первым шагом является измельчение образца и выделение ДНК. В случае костных остатков используют пескоструйные пистолеты или специализированные дрели для первичной фрагментации. Далее частицы ещё более измельчаются (до порошкообразного состояния) на перемешивающих мельницах. Порошок последовательно обрабатывается набором реагентов и подвергается центрифугированию для очистки ДНК от минералов и прочих примесей[16]. Следующий этап — изготовление ДНК-библиотек и их обогащение путем гибридизации[17]. Непосредственное получение нуклеотидной последовательности осуществляется путем секвенирования нового поколения. В 2012 году в статье журнала Methods of Molecular Biology были представлены возможные методы выделения древней ДНК из сохранившихся растительных образцов. Как и в случае с древней ДНК животных, палеорастительные образцы доходят в сохранном виде до нашего времени зачастую из-за охлаждения во время масштабных оледенений[18]. «Допотопная» ДНК «Допотопной» (англ. antediluvian) была названа ДНК старше 1 миллиона лет. Название было предложено в 1993 году Томасом Линдалом (Tomas Lindahl) в обзоре в Nature[19]. В 1990-е годы появились сообщения о секвенировании ДНК, сохранившейся на протяжении миллионов лет в ископаемых остатках растений, костях динозавров и инклюзах в янтаре. Некоторые из этих результатов с большой вероятностью являются следствием контаминации, другие оказались невоспроизводимы.[10][5] Например, в ответ на публикацию в Science о секвенировании фрагмента митохондриальной ДНК из кости динозавра мелового периода (80 млн лет назад), вышли заметки, в одной из которых было указано, что при построении филогенетического дерева последовательность из кости динозавра кластеризуется с человеческой, а не с ортологичным участком из мтДНК птиц или крокодила, что свидетельствует о большой вероятности контаминации[20]; в другой заметке высказывалось предположение[21], что последовательность, приписанная динозавру, — это на самом деле древняя вставка мтДНК в человеческой хромосоме, попавшей в образец[22]. ДНК животных плейстоцена Хорошо сохранившиеся образцы позволяют частично, а в некоторых случаях даже полностью секвенировать ядерный геном. В 2008 году была секвенирована ДНК, полученная из шерсти двух мамонтов, умерших около 20 и 59 тысяч лет назад. Картирование на черновую сборку генома африканского слона позволило оценить долю эндогенной ДНК в этих образцах в 90 % и 58 % соответственно; большую часть загрязнения в обоих случаях составили бактериальная ДНК и последовательности, происхождение которых определить не удалось. Полученные данные позволили оценить время расхождения между мамонтом и африканским слоном в 7,5 млн лет, а время расхождения между двумя линиями мамонта, для которых были взяты образцы, — в 1,5-2 млн лет. При этом секвенированная ДНК мамонтов совпадает со слоновьей на 99,41 % на нуклеотидном уровне и 99,78 % на аминокислотном уровне (то есть различие составляет примерно 1 остаток на белок). Принадлежность М4 и М25 к разным кладам была установлена ранее на основе секвенирования митохондриальной ДНК, и новая оценка на время расхождения согласуется с оценкой по мтДНК и уточняет последнюю. Одной из целей секвенирования мамонта было определение функционально важных аминокислотных различий между мамонтом и слоном. Были отобраны 92 различия между этими видами, которые могут иметь функциональное значение, и часть из которых, возможно, находилась под положительным отбором[23]. В одном из более ранних исследований в качестве образцов были взяты хорошо сохранившиеся при низких температурах останки восьми мамонтов из Музея мамонта в Хатанге. Для лучшего из отобранных образцов 45,4 % фрагментов ДНК выравнивались на сборку генома слона, при этом сходство между ДНК слона и мамонта составило 98,55 % без поправки на увеличение числа различий из-за дезаминации[24]. В 2013 году была получена черновая версия генома древней лошади[25]. Возраст образца оценивается в 560—780 тысяч лет. По состоянию на конец 2013 года это самый древний полный ядерный геном. Также были секвенированы ДНК лошади из позднего плейстоцена (~43 тысячи лет назад), пяти современных пород лошадей, лошади Пржевальского и осла. Филогенетический анализ показал, что последний общий предок всего рода Лошади жил 4-4,5 миллиона лет назад, что оказалось вдвое больше принятой до того оценки; популяции предков лошади Пржевальского и домашней лошади разошлись 38-72 тысячи лет назад. В том же году была восстановлена последовательность митохондриальной ДНК пещерного медведя из испанской Пещеры Костей, жившего в среднем плейстоцене, 179—680 тыс. лет назад, причём техника подготовки древней ДНК к секвенированию была оптимизирована для лучшего прочтения коротких (30-50 пн) фрагментов[11]. По состоянию на октябрь 2013-го, древний геном отсеквенирован с покрытием больше единицы только для таких позвоночных, как человек, белый медведь и лошадь. ДНК древних людей Неандертальцы Первым шагом в изучении генетического материала неандертальцев была работа с митохондриальной ДНК, выделенной из костей, которые были обнаружены в 1856 году в долине реки Неандерталь (Германия). В 2006 году был начат проект по секвенированию полного генома неандертальца. В работе использовалась ДНК останков из пещеры Виндия в Хорватии, а также из некоторых других костей. Выравнивание полученного генома вместе с геномом современного человека и геномом шимпанзе, позволило грубо оценить время расхождения современного человека и неандертальца в 270—440 тыс. лет в предположении, что человек и шимпанзе разошлись 6,5 млн лет назад[26]. Геномы неандертальцев из пещеры Сидрон (англ.) (Испания), пещеры Фельдхофер (англ.) (Германия), Мезмайской пещеры (Россия) незначительно отличаются от первого. Секвенирование ДНК древних людей даёт надежду установить функциональные геномные мутации, которые отличают древнего человека от обезьяны, а также проследить отличия современного человека от древних людей. Исследование выявило удивительно малое количество зафиксировавшихся замен в геноме за довольно долгое время. Было найдено всего лишь 5 генов, в которых у современного человека зафиксировалось больше одной замены, меняющей структуру белка (по сравнению с неандертальцем, который в этих локусах совпал с шимпанзе): RPTN (англ.), SPAG17, CAN15, TTF1 (англ.) и PCD16. Сравнение разнообразия SNP для неандертальцев и современного человека позволяет локализовать мишени положительного отбора в геноме. Самый обширный такой участок генома, выявленный анализом различий SNP, принадлежит гену THADA (англ.), и SNP в ближайшем его окружении ассоциированы с диабетом 2 типа, а экспрессия этого гена существенно различается для больных диабетом и здоровых людей. В этом же регионе была обнаружена вставка 9 нуклеотидов в геноме человека, которая отсутствует у всех известных геномов от мыши до приматов и у неандертальца. Также SNP в других регионах ассоциируются с шизофренией, синдромом Дауна, аутизмом. Интерес представляет ген RUNX2 (англ.), который является единственным известным на данный момент геном, связанным с развитием ключично-черепного дизостоза. Анализ SNP также выявляет примесь генетического материала неандертальцев в ДНК неафриканцев, что подтверждает теорию о скрещивании неандертальцев с современными людьми, произошедшем после выхода популяции последних из Африки. Денисовцы Именно анализ древней ДНК позволил обнаружить этот вид древних людей — изначально считалось, что найденные в Денисовой пещере фрагменты скелета (две фаланги пальцев и три коренных зуба) принадлежат неандертальцам. Секвенирование митохондриальной ДНК останков опровергло теорию и показало, что они принадлежат отдельной группе. Различие денисовцев и современных людей в 2 раза превышает различие неандертальцев и современных людей, однако для ядерной ДНК эти различия имеют одинаковый порядок. Возможное объяснение состоит в том, что денисовцы и неандертальцы происходят от общего предка, который ранее отделился от ветви будущего современного человека. Эту гипотезу подтвердило сравнение двух геномов древних людей (денисовец, неандерталец) и пяти геномов современных людей (представителей Франции, Китая, Папуа — Новой Гвинеи, африканских народностей йоруба и бушменов), а также выравнивание геномов денисовцев, неандертальцев и африканцев йоруба на геном шимпанзе. Помимо этого, исследование показало, что денисовцы и неандертальцы являются сестринскими группами. Также было показано, что денисовцы, как и неандертальцы, скрещивались с некоторыми неафриканскими популяциями современных людей: примесь ДНК неандертальцев присутствует во всех неафриканских группах, а примесь денисовцев — у папуасов и меланезийцев[14]. Филогенетические выводы по данным ядерной и митохондриальной ДНК расходятся. Это может объясняться, тем, что мтДНК денисовцев происходит из некой древней линии, которая не прижилась в неандертальцах и современных людях. Большой размер древних популяций делает эту гипотезу правдоподобной, хотя для однозначного разрешения этой проблемы данных пока недостаточно. Гейдельбергский человек Анализ практически полного митохондриального генома гейдельбергского человека из Пещеры Костей позволил оценить возраст находки в 150—640 тысяч лет и показал, что митохондриальные геномы более схожи для гейдельбергского человека и денисовцев, чем для указанных видов и неандертальца, хотя по морфологическим признакам люди из Пещеры Костей близки к неандертальцам. Есть несколько возможных объяснений. Люди из Пещеры Костей могут представлять группу, отличную и от неандертальцев, и от денисовцев, которая внесла вклад в денисовскую мтДНК, но эта версия не объясняет морфологических черт неандертальцев у вида, напрямую с ними не связанного. Второе возможное объяснение состоит в том, что гейдельбергские люди относятся к общим предкам неандертальцев и денисовцев, однако эта версия предполагает существование в этой группе двух сильно разошедшихся линий мтДНК, предковых для неандертальцев и денисовцев. В третьих, не исключено влияние малоисследованных подвидов человека, которые могли сделать вклад в мтДНК гейдельберских людей и денисовцев. Анализ ядерной ДНК может прояснить картину[27]. Древняя эпигенетика Информация о метилировании цитозинов в CpG (англ.) может быть восстановлена непосредственно по данным секвенирования[28]. При дезаминировании, которое происходит случайно с течением времени, неметилированный цитозин превращается в урацил, а метилированный — в тимин. Протокол секвенирования древней ДНК предполагает обработку ДНК урацил-ДНК-гликозилазой и эндонуклеазой VIII, в результате которой урацил удаляется и молекула ДНК раскусывается в соответствующем месте с удалением поврежденного нуклеотида[29]. В результате при секвенировании для тех позиций, где цитозин был метилирован, будет получен большой процент чтений с Т, а неметилированный цитозин прочитается как C по тем молекулам, где дезаминирование в этом месте не произошло. По последовательности ДНК, извлечённой из волос 4000-летних останков палеоэскимоса удалось построить полногеномную карту расположения нуклеосом и метилирования[30], причём профиль метилирования оказался близок к тому, что наблюдается в волосах современного человека. Также восстановлена карта метилирования для неандертальцев и денисовцев[28][31]. Их метилом оказался похож на метилом современного человека, особенно в районе генов «домашнего хозяйства», однако найдено около 2000 областей со значимыми отличиями в метилировании. В частности, у древних людей найдены заметилированные участки в кластере HOXD, задействованном в регуляции развития конечностей, что может объяснять такие анатомические различия между ними и современными людьми, как менее длинные конечности, крупные кисти, широкие локтевые и коленные суставы[28][31]. Патогены Современные методы экстракции и секвенирования древней ДНК позволяют проводить исследования патогенов, полученных из останков давно умерших от болезни людей. Филогенетический анализ полученных образцов позволяет восстанавливать эволюцию болезнетворных организмов. Секвенированы средневековые штаммы чумной палочки и палочки Хансена, а также штамм палочки Коха XIX-го века. Чумная палочка Исследования древней ДНК чумной палочки (Yersinia pestis), полученных из лондонских захоронений жертв «Чёрной смерти» установили, что чумная палочка того времени, если не считать возможных геномных перестроек, характерных для этого вида, и ограничиться однонуклеотидными полиморфизмами, мало отличалась от современных штаммов. Более того, для всех полиморфизмов, отличающих её от современного штамма, в ней присутствовал вариант предка чумной палочки — Y. pseudotuberculosis. Эти данные позволяют предполагать, что генотип древней чумной палочки не являлся главной причиной её высокой вирулентности в то время и, возможно, его вклад был наравне с вкладом таких факторов, как генетически обусловленная восприимчивость носителей, климат, социальные условия, взаимодействия с другими болезнями. При помощи филогенетического анализа был установлен временной интервал, в который жил последний общий предок всех современных патогенных для человека штаммов чумной палочки: 1282—1343 года, причём древняя бактерия оказалась ближе всего к предковому узлу филогенетического дерева[32]. Фитофтора В 2013 году было опубликовано показательное исследование в области секвенирования старых патогенов, посвященное не существующим ныне штаммам фитофторы (Phytophthora infestans), которые вызвали Ирландский картофельный голод в середине XIX века. Генетический материал этих патогенов был выделен из сохраненных листьев картофеля и томата разного времени. До настоящего времени, ввиду отсутствия методов секвенирования, анализ данных был невозможен. Ученые провели анализ разных штаммов патогенов, выделив родственные штаммы из растений в Ирландии, Северной Америки и Европы. Исследования показали, что группа штаммов вызвавшая вышеупомянутый голод (HERB-1) и Северо-Американская группа (US-1) имели последнего общего предка предположительно на территории Мексики на рубеже XVIII и XIX веков. Это исследование особенно интересно с точки зрения подходов к анализу древней ДНК возбудителей прошедших эпидемий как животных, так и растений. Данная область находится на раннем этапе своего развития, однако в случае систематического создания биологических банков, подобные знания могут значительно способствовать ускоренному поиску противомикробных препаратов в ближайшем будущем, в случае возникновения эпидемий, вызванных родственными возбудителями[33]. См. также Примечания - ↑ 1 2 3 4 B. Shapiro, M. Hofreiter (24 January 2014). «A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA». Science 343 no. 6169: 3—16. DOI:10.1126/science.1236573.
- ↑ Higuchi R. et al. DNA sequences from the quagga, an extinct member of the horse family (англ.) // Nature. — 1985. — Vol. 312, no. 5991. — P. 282—284. — DOI:10.1038/312282a0. — PMID 6504142.
- ↑ Pääbo S. Molecular cloning of Ancient Egyptian mummy DNA (англ.) // Nature. — 1985. — Vol. 314, no. 6062. — P. 644—645. — DOI:10.1038/314644a0. — PMID 3990798.
- ↑ Pääbo S. Molecular genetic investigations of ancient human remains (англ.) // Cold Spring Harb Symp Quant Biol. — 1986. — Vol. 51, no. Pt 1. — P. 441—446. — PMID 3107879.
- ↑ 1 2 3 4 5 6 7 8 9 Eske Willerslev and Alan Cooper (January 2005). «Review Paper. Ancient DNA». Proc. R. Soc. B 272 no. 1558: 3—16. DOI:10.1098/rspb.2004.2813.
- ↑ Ermanno Rizzi, Martina Lari, Elena Gigli, Gianluca De Bellis, David Caramelli Ancient DNA studies: new perspectives on old samples (En) // Genetics Selection Evolution. — 2012/12. — Т. 44, вып. 1. — С. 21. — ISSN 1297-9686. — DOI:10.1186/1297-9686-44-21.
- ↑ Pontus Skoglund, Jan Storå, Anders Götherström, Mattias Jakobsson Accurate sex identification of ancient human remains using DNA shotgun sequencing // Journal of Archaeological Science. — Т. 40, вып. 12. — С. 4477–4482. — DOI:10.1016/j.jas.2013.07.004.
- ↑ Adrian M. Whatmore Ancient-Pathogen Genomics: Coming of Age? (англ.) // mBio. — 2014-10-31. — Vol. 5, iss. 5. — P. e01676–14. — ISSN 2150-7511. — DOI:10.1128/mbio.01676-14.
- ↑ Zoe Patterson Ross, Jennifer Klunk, Gino Fornaciari, Valentina Giuffra, Sebastian Duchêne The paradox of HBV evolution as revealed from a 16th century mummy (англ.) // PLOS Pathogens. — 2018-01-04. — Vol. 14, iss. 1. — P. e1006750. — ISSN 1553-7374. — DOI:10.1371/journal.ppat.1006750.
- ↑ 1 2 3 4 Michael Hofreiter, David Serre, Hendrik N. Poinar, Melanie Kuch and Svante Pääbo (2001). «Ancient DNA». Nature Reviews Genetics 2: 353—359. DOI:10.1038/35072071.
- ↑ 1 2 Jesse Dabney et al. (06.08.2013). «Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments». PNAS (201314445): 15758—15763. DOI:10.1073/pnas.1314445110. ISSN 0027-8424.
- ↑ Pontus Skoglund, Bernd H. Northoff, Michael V. Shunkov, Anatoli P. Derevianko, Svante Pääbo, Johannes Krause, and Mattias Jakobsson. (11 February 2014). «Separating endogenous ancient DNA from modern day contamination in a Siberian Neandertal». Proc. Natl. Acad. Sci. USA vol. 111 no. 6: 2229—2234. DOI:10.1073/pnas.1318934111.
- ↑ Этот метод требует наличия референсного генома. В первом приближении метод выглядит следующим образом. Чтения выравниваются на геном, после чего рассматриваются позиции, где встречаются, например, цитозин и тимин. Сравниваются две модели: одна предполагает, что замена цитозина на тимин произошла в результате посмертной деградации ДНК, вторая предполагает, что это полиморфизм или ошибка секвенирования. Модель выбирается по методу максимального правдоподобия.
- ↑ 1 2 David Reich et al. (23/30 December 2010). «Genetic history of an archaic hominin group from Denisova Cave in Siberia». Nature vol. 468: 1053—1060. DOI:10.1038/nature09710.
- ↑ Henrik B. Hansen, Peter B. Damgaard, Ashot Margaryan, Jesper Stenderup, Niels Lynnerup Comparing Ancient DNA Preservation in Petrous Bone and Tooth Cementum (англ.) // PLOS ONE. — 2017-01-27. — Vol. 12, iss. 1. — P. e0170940. — ISSN 1932-6203. — DOI:10.1371/journal.pone.0170940.
- ↑ Jesse Dabney, Michael Knapp, Isabelle Glocke, Marie-Theres Gansauge, Antje Weihmann Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments (англ.) // Proceedings of the National Academy of Sciences. — 2013-09-24. — Vol. 110, iss. 39. — P. 15758–15763. — ISSN 1091-6490 0027-8424, 1091-6490. — DOI:10.1073/pnas.1314445110.
- ↑ Iosif Lazaridis, Dani Nadel, Gary Rollefson, Deborah C. Merrett, Nadin Rohland Genomic insights into the origin of farming in the ancient Near East (En) // Nature. — 2016/08. — Т. 536, вып. 7617. — С. 419–424. — ISSN 1476-4687. — DOI:10.1038/nature19310.
- ↑ Beth Shapiro and Michael Hofreiter (eds.). Ancient DNA: Methods and Protocols. — Methods in Molecular Biology. — © Springer Science+Business Media, LLC 2012. — С. vol. 840. — ISBN DOI 10.1007/978-1-61779-516-9_10.
- ↑ Lindahl T. Instability and decay of the primary structure of DNA (англ.) // Nature. — 1993. — Vol. 362, no. 6422. — P. 709—715. — DOI:10.1038/362709a0. — PMID 8469282.
- ↑ S. Blair Hedges, Mary H. Schweitzer (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1191—1192. DOI:10.1126/science.7761839.
- ↑ В то время сборки человеческого генома ещё не существовало. См. Проект «Геном человека»
- ↑ Zischler H., Höss M., Handt O., von Haeseler A., van der Kuyl A.C., Goudsmit J. (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1193—4. DOI:10.1126/science.7605504.
- ↑ Web Miller et al. (20 november 2008). «Sequencing the nuclear genome of the extinct woolly mammoth». Nature vol. 456: 387—392. DOI:10.1038/nature07446.
- ↑ Hendrik N. Poinar et al. (20 January 2006). «Metagenomics to Paleogenomics: Large-Scale Sequencing of Mammoth DNA». Science vol. 311: 392—394. DOI:10.1126/science.1123360.
- ↑ Ludovic Orlando et al. (26 June 2013). «Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse». Nature vol. 499: 74—78. DOI:10.1038/nature12323.
- ↑ Richard E. Green et al. (7 May 2010). «A Draft Sequence of the Neandertal Genome». Science vol. 328: 710—722. DOI:10.1126/science.1188021.
- ↑ Matthias Meyer et al. (16 January 2014). «A mitochondrial genome sequence of a hominin from Sima de los Huesos». Nature 505: 403—406. DOI:10.1038/nature12788.
- ↑ 1 2 3 Elizabeth Pennisi (18 April 2014). «Ancient DNA Holds Clues to Gene Activity in Extinct Humans». Science Vol. 344 no. 6181: pp. 245-246. DOI:10.1126/science.344.6181.245.
- ↑ Briggs et al. (22 Dec 2009). «Removal of deaminated cytosines and detection of in vivo methylation in ancient DNA». Nucl. Acids Res. Vol. 38 (6): pp. e87. DOI:10.1093/nar/gkp1163.
- ↑ Jakob Skou Pedersen et al. (3 December 2013). «Genome-wide nucleosome map and cytosine methylation levels of an ancient human genome». Genome Res 24: 454—466. DOI:10.1101/gr.163592.113.
- ↑ 1 2 David Gokhman, Eitan Lavi, Kay Prüfer, Mario F. Fraga, José A. Riancho, Janet Kelso, Svante Pääbo, Eran Meshorer, Liran Carmel (April 17 2014). «Reconstructing the DNA Methylation Maps of the Neandertal and the Denisovan». Science Published Online. DOI:10.1126/science.1250368.
- ↑ Kirsten I. Bos, Verena J. Schuenemann, G. Brian Golding, Hernán A. Burbano, Nicholas Waglechner A draft genome of Yersinia pestis from victims of the Black Death (En) // Nature. — 2011/10. — Т. 478, вып. 7370. — С. 506–510. — ISSN 1476-4687. — DOI:10.1038/nature10549.
- ↑ Kentaro Yoshida Verena J Schuenemann Liliana M Cano Marina Pais Bagdevi Mishra Rahul Sharma Chirsta Lanz Frank N Martin Sophien Kamoun Johannes Krause Marco Thines Detlef Weigel Hernán A Burbano The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine (англ.) // eLife : Published online. — 2013. — Май.
Литература wikiredia.ru Наука: Наука и техника: Lenta.ruУченые смогли расшифровать ядерную ДНК человека из останков, обнаруженных в испанском могильнике Расщелина костей (Sima de los Huesos). Оказалось, что захороненные там люди, жившие 430 тысяч лет назад, были ближе к неандертальцам, чем к денисовцам, — притом, что в их ДНК нашли следы обеих групп древних людей. Новое исследование представлено в журнале Nature. Обнаруженные в Расщелине костей в горах Сьерра-де-Атапуэрка кости изначально классифицировали как относящиеся к гейдельбергскому человеку (жил в Евразии от 600 до 250 тысяч лет назад). Этот вид считается предком неандертальцев, а некоторые исследователи относят его к предкам и современных людей. Однако в 2013 году митохондриальная ДНК (мтДНК) останков показала много общего с мтДНК денисовцев — другого вида людей, о которых антропологам почти ничего не известно (кроме того, что жили они на востоке и юго-востоке Евразии). Материалы по теме09:06 — 20 июня 2014  Главное — зубыЧерепа из Атапуэрки пролили свет на эволюцию неандертальцев 08:48 — 6 июня 2015  Палеогенетики из Германии смогли секвенировать ядерную ДНК: они извлекли достаточно материала из зуба и одной из костей ноги — несмотря на то, что древняя ДНК разложилась на короткие фрагменты. Оказалось, что гоминины из Расщелины костей ближе к неандертальцам, чем к денисовцам. Таким образом, эти группы Homo разошлись уже 430 тысяч лет назад — значительно раньше, чем считали генетики. Кроме того, это открытие отодвигает в прошлое срок отделения предков Homo sapiens от неандертальцев и денисовцев — до 765 тысяч лет назад. Ранее ученые полагали, что это событие произошло в промежутке 315-540 тысяч лет назад. Однако данные испанской ДНК, помимо всего прочего, лучше согласуются с последними датировками ископаемых останков древних людей. На следующем этапе открытие немецких палеогенетиков приведет к перечерчиванию всего эволюционного древа рода Homo. В настоящее время во всех учебниках написано, что Homo sapiens, неандертальцы и денисовцы произошли от гейдельбергского человека. Однако последний оформился в отдельный вид лишь 700 тысяч лет назад. Теперь получается, что последний общий предок трех видов — это не Homo heidelbergensis, а более древний и загадочный вид Homo antecessor, который впервые появляется более миллиона лет назад. Ученые отмечают, что останки из Расщелины костей еще не раскрыли всех своих тайн. Например, митохондриальная ДНК у них ближе к денисовской. Это можно объяснить наличием неизвестной ветви евразийских гоминин, которые скрещивались с общими предками неандертальцев и денисовцев — но не с представителями группы, которые впоследствии стали чистыми неандертальцами. Или же «испанская» мтДНК была характерна для всех этих архаичных гоминин, и лишь затем неандертальцы получили иную мтДНК из африканской популяции предков Homo sapiens. lenta.ru Секвенирование древней ДНК — википедия орг ДНК, выделенная из древнеегипетской мумии возрастом 4,000 лет ДНК, выделенная из древнеегипетской мумии возрастом 4,000 лет Секвенирование древней ДНК (aDNA, дДНК) — это секвенирование ДНК, извлечённой из древних биологических образцов, таких как палеонтологические и археологические находки, мумифицированные останки, засохшие остатки растений, копролиты. Исследования нуклеотидных последовательностей, полученных путем секвенирования древней ДНК, позволяют устанавливать филогенетические отношения между видами и проверять гипотезы о связи изменений в окружающей среде и эволюционных изменений популяций, а также предоставляют информацию для калибровки молекулярных часов[1]. При работе с древней ДНК исследователи сталкиваются со множеством проблем, связанных с сохранностью образцов. ДНК может деградировать с течением времени, химически модифицироваться. Такие методы как секвенирование нового поколения и обогащение ДНК-библиотек путем гибридизации позволяют заметно увеличить количество получаемых из образцов информации. В настоящее время возможно извлечение и анализ ДНК таких давно вымерших биологических видов, как, например, мамонты, неандертальцы или древние патогенные бактерии. История Исследования древней ДНК начались в 1984 году с секвенирования фрагмента митохондриальной ДНК квагги[2], подвида бурчелловой зебры, вымершего во второй половине XIX века. Было обнаружено, что ДНК не только сохраняется на протяжении более полутора века, но также может быть частично выделена и секвенирована. Вскоре после этого Сванте Паабо секвенировал образцы, полученные из человеческих мумий.[3][4] Эти исследования использовали бактериальное клонирование для амплификации фрагментов ДНК. Оказалось, что большая часть ДНК в образцах имеет бактериальное или грибное происхождение, а поддающаяся амплификации эндогенная ДНК состоит в основном из коротких повреждённых фрагментов многокопийных локусов (например, мтДНК) и составляет малую часть исследованной ДНК. Изобретение ПЦР позволило амплифицировать даже немногочисленные сохранившиеся фрагменты ДНК и дало толчок к развитию этой области, однако также увеличило чувствительность результатов к контаминации[5]. Области применения Исследование древней ДНК имеет важное значение для таких областей науки как генетика, палеозоология, палеоэпидемология, антропология (особенно палеоантропология) и археология. От последней развилась отдельная отрасль науки — палеогенетика. Анализ древней ДНК может использоваться для оценки эволюционной близости таксономических групп давно вымерших или сильно изменившихся организмов, конретные родственные связи которых выяснить иными способами крайне затруднительно. Особое внимание здесь уделяется областям высокой изменчивости — участкам ДНК, где мутации происходят часто. В частности, это короткие тандемные повторы (STR) и однонуклеотидные полиморфизмы (SNP). Особенно перспективным является анализ митохондриальной ДНК, так как она имеет гораздо большее количество копий по сравнению с ядерной ДНК (около 1000 копий митохондриальных ДНК и 2 копии ядерной ДНК на клетку)[6]. Анализ древней ДНК также позволяет устанавливать половую принадлежность скелетных останков представителей видов, женские и мужские особи которых отличаются набором половых хромосом, что особенно важно, когда другие методы, например антропометрия, не могут дать точного ответа[7]. Значимый интерес вызывает дДНК и в контексте палеоэпидемологии. Диагностику заболеваний по скелетным останкам и изучение древних пандемий крайне сложно проводить без анализа ДНК возбудителей, сохранившейся в останках больных. Такие исследования стали возможны только в 1990 годах. В ближайшие годы основное внимание будет уделяться обнаружению возможных вымерших инфекционных бактериальных штаммов, а также сопоставлению детектируемых по останкам симптомов болезни с современными знаниями о конкретном заболевании. С увеличением количества данных изучение дДНК может внести важный вклад в историческую эпидемиологию в будущем[8]. Технические сложности Деградация ДНК Обычно после смерти организма ДНК расщепляется эндогенными нуклеазами. Этого не происходит, если нуклеазы оказываются быстро разрушены или инактивированы, например, вследствие обезвоживания останков, низких температур или большой концентрации соли.[9] Даже в этом случае ДНК со временем повреждается в результате случайного гидролиза или окисления. К гидролитическим повреждениям относятся разрушение фосфатного остова цепи, депуринизация (англ.) (соответствующая позиция остается без азотистого основания) и дезаминирование. Чаще происходит дезаминирование цитозина в урацил, метилированный цитозин (5-метил-цитозин) дезаминируется в тимин; реже аденин превращается в гипоксантин, который комплементарен цитозину, а не тимину, что ведет к неправильному прочтению при секвенировании. Эти повреждения происходят и в живых клетках, однако там они устраняются в процессе репарации. Кроме того, возникают поперечные сшивки между цепями спирали ДНК из-за алкилирования или сшивки ДНК с различными молекулами в результате реакции Майяра. Как и разрывы цепи, поперечные сшивки мешают амплификации при ПЦР[9][5]. Перед секвенированием древнюю ДНК подвергают специальной обработке, чтобы удалить продукты дезаминирования и поперечные сшивки. Средняя длина амплифицируемых фрагментов древней ДНК часто не превосходит 100 п.н., причём для находок из одного места раскопок средняя длина фрагментов убывает с увеличением возраста находки. Современные протоколы секвенирования дДНК учитывают эту особенность; в частности, вместо отжига праймеров на фрагменты ДНК к концам фрагментов присоединяются адаптеры, а праймерами служат олигонуклеотиды, комплементарные адаптерам[10]. При низких температурах деградация ДНК идет медленнее, что обеспечивает хорошую сохранность ДНК в образцах, найденных в районе вечной мерзлоты. Однако считается, что даже в идеальных условиях ДНК не может сохраняться на протяжении более 1 млн лет[9][5]. Примесь чужеродной ДНК Часто вследствие загрязнения, или контаминации, лишь малая доля ДНК в образце имеет эндогенное происхождение. Лучшие в этом отношении образцы содержат до 90 % эндогенной ДНК.[1] Археологические образцы всегда содержат некоторое количество ДНК бактерий и грибов, которые колонизируют останки, пока те лежат в земле. Кроме того, в процессе исследования древней ДНК в образец может попасть человеческая или микробная ДНК, присутствующая в любой лаборатории. В отличие от древней ДНК современная хорошо амплифицируется при ПЦР. Продукты ПЦР, содержащие амплифицированную современную ДНК, могут распространиться по лаборатории и увеличить таким образом степень контаминации[1][5]. Для профилактики лабораторной контаминации рекомендуется продолжительная обработка оборудования ультрафиолетом или кислотой и соблюдение требований протокола работы в ПЦР-лабораториях. Исследование древней ДНК не должно проводиться в лаборатории, где ранее изучались современные образцы, особенно близких видов. Хорошим способом убедиться (и убедить научное сообщество) в истинности результатов является повторение исследования в другой лаборатории[5]. Многие археологические образцы, особенно найденные давно, подверглись контаминации в процессе извлечения и исследования. Если образец был найден несколько десятилетий назад, человеческая ДНК, которой он загрязнен, может обладать всеми признаками древней ДНК. Кости и зубы имеют пористую поверхность, что делает проблематичной их очистку от современной ДНК. Волосы оказываются в этом отношении предпочтительнее: содержащаяся в них ДНК малодоступна для бактерий, а гидрофобная поверхность позволяет очистить их перед извлечением ДНК[1]. В этом случае совпадение последовательностей, полученных в результате независимого исследования разных частей образца (например, бедра и зубов) в разных лабораториях свидетельствует в пользу истинности результата[5]. В случае человеческих останков контаминация современной человеческой ДНК может приводить к ошибкам при исследовании филогении и популяционной генетики. Существует статистический метод, позволяющий отличать древнюю ДНК от современной по паттерну дезаминирования[11]. Если известно, что образец ДНК принадлежит женщине, можно картировать отсеквенированные последовательности на Y-хромосому и детектировать таким образом попадание в образец мужской ДНК[12]. Если исследуется ДНК нечеловеческого происхождения, очевидным способом проверки на контаминацию является картирование полученных при секвенировании чтений на человеческий геном, а также на геномы других организмов в случае, если они могли быть источниками контаминации. При секвенировании древней бактериальной ДНК проблема состоит также в том, что известны геномы далеко не всех существующих ныне бактерий, так что из того, что полученная последовательность не входит в известные бактериальные геномы, нельзя заключить, что она принадлежит древней бактерии, а не получена в результате контаминации[5]. Вставки мтДНК и хпДНК в хромосомах Вставки митохондриальной и пластидной ДНК часто встречаются в ядерной ДНК. При исследовании древних образцов ДНК нельзя выделить ДНК органелл, поэтому эти вставки могут быть источником ошибок, так как их не всегда легко отличить по последовательности от настоящей мтДНК или хпДНК. Если такие вставки будут приняты за ДНК органелл, это может исказить результаты исследования филогении или популяционной генетики[5]. Методы обработки древней ДНК В качестве источника древней ДНК часто стараются использовать сохранившиеся части тела, не представляющие большого интереса с точки зрения анатомического строения. Из-за описанных выше процессов в большинстве древних образцов содержание интересующей исследователей древней ДНК очень мало — всего около 1 %. Это количество сильно варьирует в зависимости от природы образца. Последние исследования показывают, что значительно больший выход эндогенной ДНК из останков предков человека достигается при выделении материала из зубов и каменистой части височной кости.[13] Первым шагом является измельчение образца и выделение ДНК. В случае костных остатков используют пескоструйные пистолеты или специализированные дрели для первичной фрагментации. Далее частицы ещё более измельчаются(до порошкообразного состояния) на перемешивающих мельницах. Порошок последовательно обрабатывается набором реагентов и подвергается центрифугированию для очистки ДНК от минералов и прочих примесей[14]. Следующий этап — изготовление ДНК-библиотек и их обогащение путем гибридизации[15]. Непосредственное получение нуклеотидной последовательности осуществляется путем секвенирования нового поколения. В 2012 году в статье журнала Methods of Molecular Biology были представлены возможные методы выделения древней ДНК из сохранившихся растительных образцов. Как и в случае с древней ДНК животных, палеорастительные образцы доходят в сохранном виде до нашего времени зачастую из-за охлаждения во время масштабных оледенений[16]. «Допотопная» ДНК «Допотопной» (англ. antediluvian) была названа ДНК старше 1 миллиона лет. Название было предложено в 1993 году Томасом Линдалом (Tomas Lindahl) в обзоре в Nature[17]. В 90-е годы появились сообщения о секвенировании ДНК, сохранившейся на протяжении миллионов лет в ископаемых остатках растений, костях динозавров и инклюзах в янтаре. Некоторые из этих результатов с большой вероятностью являются следствием контаминации, другие оказались невоспроизводимы.[9][5] Например, в ответ на публикацию в Science о секвенировании фрагмента митохондриальной ДНК из кости динозавра мелового периода (80 млн лет назад), вышли заметки, в одной из которых было указано, что при построении филогенетического дерева последовательность из кости динозавра кластеризуется с человеческой, а не с ортологичным участком из мтДНК птиц или крокодила, что свидетельствует о большой вероятности контаминации[18]; в другой заметке высказывалось предположение[19], что последовательность, приписанная динозавру, — это на самом деле древняя вставка мтДНК в человеческой хромосоме, попавшей в образец[20]. ДНК животных плейстоцена Хорошо сохранившиеся образцы позволяют частично, а в некоторых случаях даже полностью секвенировать ядерный геном. В 2008 году была секвенирована ДНК, полученная из шерсти двух мамонтов, умерших около 20 и 59 тысяч лет назад. Картирование на черновую сборку генома африканского слона позволило оценить долю эндогенной ДНК в этих образцах в 90 % и 58 % соответственно; большую часть загрязнения в обоих случаях составили бактериальная ДНК и последовательности, происхождение которых определить не удалось. Полученные данные позволили оценить время расхождения между мамонтом и африканским слоном в 7,5 млн лет, а время расхождения между двумя линиями мамонта, для которых были взяты образцы, — в 1,5-2 млн лет. При этом секвенированная ДНК мамонтов совпадает со слоновьей на 99,41 % на нуклеотидном уровне и 99,78 % на аминокислотном уровне (то есть различие составляет примерно 1 остаток на белок). Принадлежность М4 и М25 к разным кладам была установлена ранее на основе секвенирования митохондриальной ДНК, и новая оценка на время расхождения согласуется с оценкой по мтДНК и уточняет последнюю. Одной из целей секвенирования мамонта было определение функционально важных аминокислотных различий между мамонтом и слоном. Были отобраны 92 различия между этими видами, которые могут иметь функциональное значение, и часть из которых, возможно, находилась под положительным отбором[21]. В одном из более ранних исследований в качестве образцов были взяты хорошо сохранившиеся при низких температурах останки восьми мамонтов из Музея мамонта в Хатанге. Для лучшего образца 45,4 % фрагментов ДНК выравнивались на сборку генома слона, при этом сходство между ДНК слона и мамонта составило 98,55 % без поправки на увеличение числа различий из-за дезаминации[22]. В 2013 году была получена черновая версия генома древней лошади[23]. Возраст образца оценивается в 560—780 тысяч лет. По состоянию на конец 2013 года это самый древний полный ядерный геном. Также были секвенированы ДНК лошади из позднего плейстоцена (~43 тысячи лет назад), пяти современных пород лошадей, лошади Пржевальского и осла. Филогенетический анализ показал, что последний общий предок всего рода Лошади жил 4-4,5 миллиона лет назад, что оказалось вдвое больше принятой до того оценки; популяции предков лошади Пржевальского и домашней лошади разошлись 38-72 тысячи лет назад. В том же году была восстановлена последовательность митохондриальной ДНК пещерного медведя из испанской Пещеры Костей, жившего в среднем плейстоцене, 179—680 тыс. лет назад, причём техника подготовки дДНК к секвенированию была оптимизирована для лучшего прочтения коротких (30-50 пн) фрагментов[10]. По состоянию на октябрь 2013-го, древний геном отсеквенирован с покрытием больше единицы только для таких позвоночных, как человек, белый медведь и лошадь. ДНК древних людей Неандертальцы На данный момент в европейской части Евразии найдено множество останков неандертальцев. Первым шагом в изучении их генетического материала была работа с митохондриальной ДНК, выделенной из костей, которые были обнаружены в 1856 году в долине реки Неандерталь (Германия). В 2006 году был начат проект по секвенированию полного генома неандертальца. В работе использовалась ДНК останков из пещеры Виндия в Хорватии, а также из некоторых других костей. Выравнивание полученного генома вместе с геномом современного человека и геномом шимпанзе, позволило грубо оценить время расхождения современного человека и неандертальца в 270—440 тыс. лет в предположении, что человек и шимпанзе разошлись 6,5 млн лет назад[24]. Геномы неандертальцев из пещеры Сидрон (англ.) (Испания), пещеры Фельдхофер (англ.) (Германия), Мезмайской пещеры (Россия) незначительно отличаются от первого. Секвенирование ДНК древних людей даёт надежду установить функциональные геномные мутации, которые отличают древнего человека от обезьяны, а также проследить отличия современного человека от древних людей. Исследование выявило удивительно малое количество зафиксировавшихся замен в геноме за довольно долгое время. Было найдено всего лишь 5 генов, в которых у современного человека зафиксировалось больше одной замены, меняющей структуру белка (по сравнению с неандертальцем, который в этих локусах совпал с шимпанзе): RPTN (англ.), SPAG17, CAN15, TTF1 (англ.) и PCD16. Сравнение разнообразия SNP для неандертальцев и современного человека позволяет локализовать мишени положительного отбора в геноме. Самый обширный такой участок генома, выявленный анализом различий SNP, принадлежит гену THADA (англ.), и SNP в ближайшем его окружении ассоциированы с диабетом 2 типа, а экспрессия этого гена существенно различается для больных диабетом и здоровых людей. В этом же регионе была обнаружена вставка 9 нуклеотидов в геноме человека, которая отсутствует у всех известных геномов от мыши до приматов и у неандертальца. Также SNP в других регионах ассоциируются с шизофренией, синдромом Дауна, аутизмом. Интерес представляет ген RUNX2 (англ.), который является единственным известным на данный момент геном, связанным с развитием ключично-черепного дизостоза. Анализ SNP также выявляет примесь генетического материала неандертальцев в ДНК неафриканцев, что подтверждает теорию о скрещивании неандертальцев с современными людьми, произошедшем после выхода популяции последних из Африки. Денисовцы Именно анализ древней ДНК позволил обнаружить этот вид древних людей — изначально считалось, что найденные в Денисовой пещере фрагменты скелета (две фаланги пальцев и три коренных зуба) принадлежат неандертальцам. Секвенирование митохондриальной ДНК останков опровергло теорию и показало, что они принадлежат отдельной группе. Различие денисовцев и современных людей в 2 раза превышает различие неандертальцев и современных людей, однако для ядерной ДНК эти различия имеют одинаковый порядок. Возможное объяснение состоит в том, что денисовцы и неандертальцы происходят от общего предка, который ранее отделился от ветви будущего современного человека. Эту гипотезу подтвердило сравнение двух геномов древних людей (денисовец, неандерталец) и пяти геномов современных людей (представителей Франции, Китая, Папуа — Новой Гвинеи, африканских народностей Йоруба и Бушменов), а также выравнивание геномов денисовцев, неандертальцев и африканцев Йоруба на геном шимпанзе. Помимо этого, исследование показало, что денисовцы и неандертальцы являются сестринскими группами. Также было показано, что денисовцы, как и неандертальцы, скрещивались с некоторыми неафриканскими популяциями современных людей: примесь ДНК неандертальцев присутствует во всех неафриканских группах, а примесь денисовцев — у папуасов и меланезийцев.[12]. Филогенетические выводы по данным ядерной и митохондриальной ДНК расходятся. Это может объясняться, тем, что мтДНК денисовцев происходит из некой древней линии, которая не прижилась в неандертальцах и современных людях. Большой размер древних популяций делает эту гипотезу правдоподобной, хотя для однозначного разрешения этой проблемы данных пока недостаточно. Гейдельбергский человек Анализ практически полного митохондриального генома гейдельбергского человека из Пещеры Костей позволил оценить возраст находки в 150—640 тысяч лет и показал, что митохондриальные геномы более схожи для гейдельбергского человека и денисовцев, чем для указанных видов и неандертальца, хотя по морфологическим признакам люди из Пещеры Костей близки к неандертальцам. Есть несколько возможных объяснений. Люди из Пещеры Костей могут представлять группу, отличную и от неандертальцев, и от денисовцев, которая внесла вклад в денисовскую мтДНК, но эта версия не объясняет морфологических черт неандертальцев у вида, напрямую с ними не связанного. Второе возможное объяснение состоит в том, что гейдельбергские люди относятся к общим предкам неандертальцев и денисовцев, однако эта версия предполагает существование в этой группе двух сильно разошедшихся линий мтДНК, предковых для неандертальцев и денисовцев. В третьих, не исключено влияние малоисследованных подвидов человека, которые могли сделать вклад в мтДНК гейдельберских людей и денисовцев. Анализ ядерной ДНК может прояснить картину[25]. Древняя эпигенетика Информация о метилировании цитозинов в CpG (англ.) может быть восстановлена непосредственно по данным секвенирования[26]. При дезаминировании, которое происходит случайно с течением времени, неметилированный цитозин превращается в урацил, а метилированный — в тимин. Протокол секвенирования древней ДНК предполагает обработку ДНК урацил-ДНК-гликозилазой и эндонуклеазой VIII, в результате которой урацил удаляется и молекула ДНК раскусывается в соответствующем месте с удалением поврежденного нуклеотида[27]. В результате при секвенировании для тех позиций, где цитозин был метилирован, будет получен большой процент чтений с Т, а неметилированный цитозин прочитается как C по тем молекулам, где дезаминирование в этом месте не произошло. По последовательности ДНК, извлечённой из волос 4000-летних останков палеоэскимоса удалось построить полногеномную карту расположения нуклеосом и метилирования[28], причём профиль метилирования оказался близок к тому, что наблюдается в волосах современного человека. Также восстановлена карта метилирования для неандертальцев и денисовцев[26][29]. Их метилом оказался похож на метилом современного человека, особенно в районе генов «домашнего хозяйства», однако найдено около 2000 областей со значимыми отличиями в метилировании. В частности, у древних людей найдены заметилированные участки в кластере HOXD, задействованном в регуляции развития конечностей, что может объяснять такие анатомические различия между ними и современными людьми, как менее длинные конечности, крупные кисти, широкие локтевые и коленные суставы[26][29]. Патогены Современные методы экстракции и секвенирования древней ДНК позволяют проводить исследования патогенов, полученных из останков давно умерших от болезни людей. Филогенетический анализ полученных образцов позволяет восстанавливать эволюцию болезнетворных организмов. Секвенированы средневековые штаммы чумной палочки и палочки Хансена, а также штамм палочки Коха XIX-го века. Чумная палочка Исследования дДНК чумной палочки (Yersinia pestis), полученных из лондонских захоронений жертв «Чёрной смерти» установили, что чумная палочка того времени, если не считать возможных геномных перестроек, характерных для этого вида, и ограничиться однонуклеотидными полиморфизмами, мало отличалась от современных штаммов. Более того, для всех полиморфизмов, отличающих её от современного штамма, в ней присутствовал вариант предка чумной палочки — Y. pseudotuberculosis. Эти данные позволяют предполагать, что генотип древней чумной палочки не являлся главной причиной её высокой вирулентности в то время и, возможно, его вклад был наравне с вкладом таких факторов, как генетически обусловленная восприимчивость носителей, климат, социальные условия, взаимодействия с другими болезнями. При помощи филогенетического анализа был установлен временной интервал, в который жил последний общий предок всех современных патогенных для человека штаммов чумной палочки: 1282—1343 года, причём древняя бактерия оказалась ближе всего к предковому узлу филогенетического дерева[30]. Фитофтора В 2013 году было опубликовано показательное исследование в области секвенирования старых патогенов, посвященное не существующим ныне штаммам фитофторы (Phytophthora infestans), которые вызвали Великий Картофельный Голод в Ирландии в середине XIX века. Генетический материал этих патогенов был выделен из сохраненных листьев картофеля и томата разного времени. До настоящего времени, ввиду отсутствия методов секвенирования, анализ данных был невозможен. Ученые провели анализ разных штаммов патогенов, выделив родственные штаммы из растений в Ирландии, Северной Америки и Европы. Исследования показали, что группа штаммов вызвавшая вышеупомянутый голод (HERB-1) и Северо-Американская группа (US-1) имели последнего общего предка предположительно на территории Мексики на рубеже XVIII и XIX веков. Это исследование особенно интересно с точки зрения подходов к анализу дДНК возбудителей прошедших эпидемий как животных, так и растений. Данная область находится на раннем этапе своего развития, однако в случае систематического создания биологических банков, подобные знания могут значительно способствовать ускоренному поиску противомикробных препаратов в ближайшем будущем, в случае возникновения эпидемий, вызванных родственными возбудителями[31]. См. такжеПримечания - ↑ 1 2 3 4 B. Shapiro, M. Hofreiter (24 January 2014). «A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA». Science 343 no. 6169: 3—16. DOI:10.1126/science.1236573.
- ↑ Higuchi R. et al. DNA sequences from the quagga, an extinct member of the horse family (англ.) // Nature. — 1985. — Vol. 312, no. 5991. — P. 282—284. — DOI:10.1038/312282a0. — PMID 6504142.
- ↑ Pääbo S. Molecular cloning of Ancient Egyptian mummy DNA (англ.) // Nature. — 1985. — Vol. 314, no. 6062. — P. 644—645. — DOI:10.1038/314644a0. — PMID 3990798.
- ↑ Pääbo S. Molecular genetic investigations of ancient human remains (англ.) // Cold Spring Harb Symp Quant Biol. — 1986. — Vol. 51, no. Pt 1. — P. 441—446. — PMID 3107879.
- ↑ 1 2 3 4 5 6 7 8 9 Eske Willerslev and Alan Cooper (January 2005). «Review Paper. Ancient DNA». Proc. R. Soc. B 272 no. 1558: 3—16. DOI:10.1098/rspb.2004.2813.
- ↑ Ermanno Rizzi, Martina Lari, Elena Gigli, Gianluca De Bellis, David Caramelli Ancient DNA studies: new perspectives on old samples (En) // Genetics Selection Evolution. — 2012/12. — Т. 44, вып. 1. — С. 21. — ISSN 1297-9686. — DOI:10.1186/1297-9686-44-21.
- ↑ Pontus Skoglund, Jan Storå, Anders Götherström, Mattias Jakobsson Accurate sex identification of ancient human remains using DNA shotgun sequencing // Journal of Archaeological Science. — Т. 40, вып. 12. — С. 4477–4482. — DOI:10.1016/j.jas.2013.07.004.
- ↑ Adrian M. Whatmore Ancient-Pathogen Genomics: Coming of Age? (англ.) // mBio. — 2014-10-31. — Vol. 5, iss. 5. — P. e01676–14. — ISSN 2150-7511. — DOI:10.1128/mbio.01676-14.
- ↑ 1 2 3 4 Michael Hofreiter, David Serre, Hendrik N. Poinar, Melanie Kuch and Svante Pääbo (2001). «Ancient DNA». Nature Reviews Genetics 2: 353—359. DOI:10.1038/35072071.
- ↑ 1 2 Jesse Dabney et al. (9 September 2013). «Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments». PNAS. DOI:10.1073/pnas.1314445110.
- ↑ Pontus Skoglund, Bernd H. Northoff, Michael V. Shunkov, Anatoli P. Derevianko, Svante Pääbo, Johannes Krause, and Mattias Jakobsson. (11 February 2014). «Separating endogenous ancient DNA from modern day contamination in a Siberian Neandertal». Proc. Natl. Acad. Sci. USA vol. 111 no. 6: 2229—2234. DOI:10.1073/pnas.1318934111. Этот метод требует наличия референсного генома. В первом приближении метод выглядит следующим образом. Чтения выравниваются на геном, после чего рассматриваются позиции, где встречаются, например, цитозин и тимин. Сравниваются две модели: одна предполагает, что замена цитозина на тимин произошла в результате посмертной деградации ДНК, вторая предполагает, что это полиморфизм или ошибка секвенирования. Модель выбирается по методу максимального правдоподобия.
- ↑ 1 2 David Reich et al. (23/30 December 2010). «Genetic history of an archaic hominin group from Denisova Cave in Siberia». Nature vol. 468: 1053—1060. DOI:10.1038/nature09710.
- ↑ Henrik B. Hansen, Peter B. Damgaard, Ashot Margaryan, Jesper Stenderup, Niels Lynnerup Comparing Ancient DNA Preservation in Petrous Bone and Tooth Cementum (англ.) // PLOS ONE. — 2017-01-27. — Vol. 12, iss. 1. — P. e0170940. — ISSN 1932-6203. — DOI:10.1371/journal.pone.0170940.
- ↑ Jesse Dabney, Michael Knapp, Isabelle Glocke, Marie-Theres Gansauge, Antje Weihmann Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments (англ.) // Proceedings of the National Academy of Sciences. — 2013-09-24. — Vol. 110, iss. 39. — P. 15758–15763. — ISSN 1091-6490 0027-8424, 1091-6490. — DOI:10.1073/pnas.1314445110.
- ↑ Iosif Lazaridis, Dani Nadel, Gary Rollefson, Deborah C. Merrett, Nadin Rohland Genomic insights into the origin of farming in the ancient Near East (En) // Nature. — 2016/08. — Т. 536, вып. 7617. — С. 419–424. — ISSN 1476-4687. — DOI:10.1038/nature19310.
- ↑ Beth Shapiro and Michael Hofreiter (eds.). Ancient DNA: Methods and Protocols. — Methods in Molecular Biology. — © Springer Science+Business Media, LLC 2012. — С. vol. 840. — ISBN DOI 10.1007/978-1-61779-516-9_10.
- ↑ Lindahl T. Instability and decay of the primary structure of DNA (англ.) // Nature. — 1993. — Vol. 362, no. 6422. — P. 709—715. — DOI:10.1038/362709a0. — PMID 8469282.
- ↑ S. Blair Hedges, Mary H. Schweitzer (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1191—1192. DOI:10.1126/science.7761839.
- ↑ В то время сборки человеческого генома ещё не существовало. См. Проект «Геном человека»
- ↑ Zischler H., Höss M., Handt O., von Haeseler A., van der Kuyl A.C., Goudsmit J. (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1193—4. DOI:10.1126/science.7605504.
- ↑ Web Miller et al. (20 november 2008). «Sequencing the nuclear genome of the extinct woolly mammoth». Nature vol. 456: 387—392. DOI:10.1038/nature07446.
- ↑ Hendrik N. Poinar et al. (20 January 2006). «Metagenomics to Paleogenomics: Large-Scale Sequencing of Mammoth DNA». Science vol. 311: 392—394. DOI:10.1126/science.1123360.
- ↑ Ludovic Orlando et al. (26 June 2013). «Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse». Nature vol. 499: 74—78. DOI:10.1038/nature12323.
- ↑ Richard E. Green et al. (7 May 2010). «A Draft Sequence of the Neandertal Genome». Science vol. 328: 710—722. DOI:10.1126/science.1188021.
- ↑ Matthias Meyer et al. (16 January 2014). «A mitochondrial genome sequence of a hominin from Sima de los Huesos». Nature 505: 403—406. DOI:10.1038/nature12788.
- ↑ 1 2 3 Elizabeth Pennisi (18 April 2014). «Ancient DNA Holds Clues to Gene Activity in Extinct Humans». Science Vol. 344 no. 6181: pp. 245-246. DOI:10.1126/science.344.6181.245.
- ↑ Briggs et al. (22 Dec 2009). «Removal of deaminated cytosines and detection of in vivo methylation in ancient DNA». Nucl. Acids Res. Vol. 38 (6): pp. e87. DOI:10.1093/nar/gkp1163.
- ↑ Jakob Skou Pedersen et al. (3 December 2013). «Genome-wide nucleosome map and cytosine methylation levels of an ancient human genome». Genome Res 24: 454—466. DOI:10.1101/gr.163592.113.
- ↑ 1 2 David Gokhman, Eitan Lavi, Kay Prüfer, Mario F. Fraga, José A. Riancho, Janet Kelso, Svante Pääbo, Eran Meshorer, Liran Carmel (April 17 2014). «Reconstructing the DNA Methylation Maps of the Neandertal and the Denisovan». Science Published Online. DOI:10.1126/science.1250368.
- ↑ Kirsten I. Bos, Verena J. Schuenemann, G. Brian Golding, Hernán A. Burbano, Nicholas Waglechner A draft genome of Yersinia pestis from victims of the Black Death (En) // Nature. — 2011/10. — Т. 478, вып. 7370. — С. 506–510. — ISSN 1476-4687. — DOI:10.1038/nature10549.
- ↑ Kentaro Yoshida Verena J Schuenemann Liliana M Cano Marina Pais Bagdevi Mishra Rahul Sharma Chirsta Lanz Frank N Martin Sophien Kamoun Johannes Krause Marco Thines Detlef Weigel Hernán A Burbano The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine (англ.) // eLife. — 2013. — Май.
Литератураwww-wikipediya.ru Секвенирование древней ДНК — Википедия РУ ДНК, выделенная из древнеегипетской мумии возрастом 4,000 лет ДНК, выделенная из древнеегипетской мумии возрастом 4,000 лет Секвенирование древней ДНК (aDNA, дДНК) — это секвенирование ДНК, извлечённой из древних биологических образцов, таких как палеонтологические и археологические находки, мумифицированные останки, засохшие остатки растений, копролиты. Исследования нуклеотидных последовательностей, полученных путем секвенирования древней ДНК, позволяют устанавливать филогенетические отношения между видами и проверять гипотезы о связи изменений в окружающей среде и эволюционных изменений популяций, а также предоставляют информацию для калибровки молекулярных часов[1]. При работе с древней ДНК исследователи сталкиваются со множеством проблем, связанных с сохранностью образцов. ДНК может деградировать с течением времени, химически модифицироваться. Такие методы как секвенирование нового поколения и обогащение ДНК-библиотек путем гибридизации позволяют заметно увеличить количество получаемых из образцов информации. В настоящее время возможно извлечение и анализ ДНК таких давно вымерших биологических видов, как, например, мамонты, неандертальцы или древние патогенные бактерии. История Исследования древней ДНК начались в 1984 году с секвенирования фрагмента митохондриальной ДНК квагги[2], подвида бурчелловой зебры, вымершего во второй половине XIX века. Было обнаружено, что ДНК не только сохраняется на протяжении более полутора века, но также может быть частично выделена и секвенирована. Вскоре после этого Сванте Паабо секвенировал образцы, полученные из человеческих мумий.[3][4] Эти исследования использовали бактериальное клонирование для амплификации фрагментов ДНК. Оказалось, что большая часть ДНК в образцах имеет бактериальное или грибное происхождение, а поддающаяся амплификации эндогенная ДНК состоит в основном из коротких повреждённых фрагментов многокопийных локусов (например, мтДНК) и составляет малую часть исследованной ДНК. Изобретение ПЦР позволило амплифицировать даже немногочисленные сохранившиеся фрагменты ДНК и дало толчок к развитию этой области, однако также увеличило чувствительность результатов к контаминации[5]. Области применения Исследование древней ДНК имеет важное значение для таких областей науки как генетика, палеозоология, палеоэпидемология, антропология (особенно палеоантропология) и археология. От последней развилась отдельная отрасль науки — палеогенетика. Анализ древней ДНК может использоваться для оценки эволюционной близости таксономических групп давно вымерших или сильно изменившихся организмов, конретные родственные связи которых выяснить иными способами крайне затруднительно. Особое внимание здесь уделяется областям высокой изменчивости — участкам ДНК, где мутации происходят часто. В частности, это короткие тандемные повторы (STR) и однонуклеотидные полиморфизмы (SNP). Особенно перспективным является анализ митохондриальной ДНК, так как она имеет гораздо большее количество копий по сравнению с ядерной ДНК (около 1000 копий митохондриальных ДНК и 2 копии ядерной ДНК на клетку)[6]. Анализ древней ДНК также позволяет устанавливать половую принадлежность скелетных останков представителей видов, женские и мужские особи которых отличаются набором половых хромосом, что особенно важно, когда другие методы, например антропометрия, не могут дать точного ответа[7]. Значимый интерес вызывает дДНК и в контексте палеоэпидемологии. Диагностику заболеваний по скелетным останкам и изучение древних пандемий крайне сложно проводить без анализа ДНК возбудителей, сохранившейся в останках больных. Такие исследования стали возможны только в 1990 годах. В ближайшие годы основное внимание будет уделяться обнаружению возможных вымерших инфекционных бактериальных штаммов, а также сопоставлению детектируемых по останкам симптомов болезни с современными знаниями о конкретном заболевании. С увеличением количества данных изучение дДНК может внести важный вклад в историческую эпидемиологию в будущем[8]. Технические сложности Деградация ДНК Обычно после смерти организма ДНК расщепляется эндогенными нуклеазами. Этого не происходит, если нуклеазы оказываются быстро разрушены или инактивированы, например, вследствие обезвоживания останков, низких температур или большой концентрации соли.[9] Даже в этом случае ДНК со временем повреждается в результате случайного гидролиза или окисления. К гидролитическим повреждениям относятся разрушение фосфатного остова цепи, депуринизация (англ.) (соответствующая позиция остается без азотистого основания) и дезаминирование. Чаще происходит дезаминирование цитозина в урацил, метилированный цитозин (5-метил-цитозин) дезаминируется в тимин; реже аденин превращается в гипоксантин, который комплементарен цитозину, а не тимину, что ведет к неправильному прочтению при секвенировании. Эти повреждения происходят и в живых клетках, однако там они устраняются в процессе репарации. Кроме того, возникают поперечные сшивки между цепями спирали ДНК из-за алкилирования или сшивки ДНК с различными молекулами в результате реакции Майяра. Как и разрывы цепи, поперечные сшивки мешают амплификации при ПЦР[9][5]. Перед секвенированием древнюю ДНК подвергают специальной обработке, чтобы удалить продукты дезаминирования и поперечные сшивки. Средняя длина амплифицируемых фрагментов древней ДНК часто не превосходит 100 п.н., причём для находок из одного места раскопок средняя длина фрагментов убывает с увеличением возраста находки. Современные протоколы секвенирования дДНК учитывают эту особенность; в частности, вместо отжига праймеров на фрагменты ДНК к концам фрагментов присоединяются адаптеры, а праймерами служат олигонуклеотиды, комплементарные адаптерам[10]. При низких температурах деградация ДНК идет медленнее, что обеспечивает хорошую сохранность ДНК в образцах, найденных в районе вечной мерзлоты. Однако считается, что даже в идеальных условиях ДНК не может сохраняться на протяжении более 1 млн лет[9][5]. Примесь чужеродной ДНК Часто вследствие загрязнения, или контаминации, лишь малая доля ДНК в образце имеет эндогенное происхождение. Лучшие в этом отношении образцы содержат до 90 % эндогенной ДНК.[1] Археологические образцы всегда содержат некоторое количество ДНК бактерий и грибов, которые колонизируют останки, пока те лежат в земле. Кроме того, в процессе исследования древней ДНК в образец может попасть человеческая или микробная ДНК, присутствующая в любой лаборатории. В отличие от древней ДНК современная хорошо амплифицируется при ПЦР. Продукты ПЦР, содержащие амплифицированную современную ДНК, могут распространиться по лаборатории и увеличить таким образом степень контаминации[1][5]. Для профилактики лабораторной контаминации рекомендуется продолжительная обработка оборудования ультрафиолетом или кислотой и соблюдение требований протокола работы в ПЦР-лабораториях. Исследование древней ДНК не должно проводиться в лаборатории, где ранее изучались современные образцы, особенно близких видов. Хорошим способом убедиться (и убедить научное сообщество) в истинности результатов является повторение исследования в другой лаборатории[5]. Многие археологические образцы, особенно найденные давно, подверглись контаминации в процессе извлечения и исследования. Если образец был найден несколько десятилетий назад, человеческая ДНК, которой он загрязнен, может обладать всеми признаками древней ДНК. Кости и зубы имеют пористую поверхность, что делает проблематичной их очистку от современной ДНК. Волосы оказываются в этом отношении предпочтительнее: содержащаяся в них ДНК малодоступна для бактерий, а гидрофобная поверхность позволяет очистить их перед извлечением ДНК[1]. В этом случае совпадение последовательностей, полученных в результате независимого исследования разных частей образца (например, бедра и зубов) в разных лабораториях свидетельствует в пользу истинности результата[5]. В случае человеческих останков контаминация современной человеческой ДНК может приводить к ошибкам при исследовании филогении и популяционной генетики. Существует статистический метод, позволяющий отличать древнюю ДНК от современной по паттерну дезаминирования[11]. Если известно, что образец ДНК принадлежит женщине, можно картировать отсеквенированные последовательности на Y-хромосому и детектировать таким образом попадание в образец мужской ДНК[12]. Если исследуется ДНК нечеловеческого происхождения, очевидным способом проверки на контаминацию является картирование полученных при секвенировании чтений на человеческий геном, а также на геномы других организмов в случае, если они могли быть источниками контаминации. При секвенировании древней бактериальной ДНК проблема состоит также в том, что известны геномы далеко не всех существующих ныне бактерий, так что из того, что полученная последовательность не входит в известные бактериальные геномы, нельзя заключить, что она принадлежит древней бактерии, а не получена в результате контаминации[5]. Вставки мтДНК и хпДНК в хромосомах Вставки митохондриальной и пластидной ДНК часто встречаются в ядерной ДНК. При исследовании древних образцов ДНК нельзя выделить ДНК органелл, поэтому эти вставки могут быть источником ошибок, так как их не всегда легко отличить по последовательности от настоящей мтДНК или хпДНК. Если такие вставки будут приняты за ДНК органелл, это может исказить результаты исследования филогении или популяционной генетики[5]. Методы обработки древней ДНК В качестве источника древней ДНК часто стараются использовать сохранившиеся части тела, не представляющие большого интереса с точки зрения анатомического строения. Из-за описанных выше процессов в большинстве древних образцов содержание интересующей исследователей древней ДНК очень мало — всего около 1 %. Это количество сильно варьирует в зависимости от природы образца. Последние исследования показывают, что значительно больший выход эндогенной ДНК из останков предков человека достигается при выделении материала из зубов и каменистой части височной кости.[13] Первым шагом является измельчение образца и выделение ДНК. В случае костных остатков используют пескоструйные пистолеты или специализированные дрели для первичной фрагментации. Далее частицы ещё более измельчаются(до порошкообразного состояния) на перемешивающих мельницах. Порошок последовательно обрабатывается набором реагентов и подвергается центрифугированию для очистки ДНК от минералов и прочих примесей[14]. Следующий этап — изготовление ДНК-библиотек и их обогащение путем гибридизации[15]. Непосредственное получение нуклеотидной последовательности осуществляется путем секвенирования нового поколения. В 2012 году в статье журнала Methods of Molecular Biology были представлены возможные методы выделения древней ДНК из сохранившихся растительных образцов. Как и в случае с древней ДНК животных, палеорастительные образцы доходят в сохранном виде до нашего времени зачастую из-за охлаждения во время масштабных оледенений[16]. «Допотопная» ДНК «Допотопной» (англ. antediluvian) была названа ДНК старше 1 миллиона лет. Название было предложено в 1993 году Томасом Линдалом (Tomas Lindahl) в обзоре в Nature[17]. В 90-е годы появились сообщения о секвенировании ДНК, сохранившейся на протяжении миллионов лет в ископаемых остатках растений, костях динозавров и инклюзах в янтаре. Некоторые из этих результатов с большой вероятностью являются следствием контаминации, другие оказались невоспроизводимы.[9][5] Например, в ответ на публикацию в Science о секвенировании фрагмента митохондриальной ДНК из кости динозавра мелового периода (80 млн лет назад), вышли заметки, в одной из которых было указано, что при построении филогенетического дерева последовательность из кости динозавра кластеризуется с человеческой, а не с ортологичным участком из мтДНК птиц или крокодила, что свидетельствует о большой вероятности контаминации[18]; в другой заметке высказывалось предположение[19], что последовательность, приписанная динозавру, — это на самом деле древняя вставка мтДНК в человеческой хромосоме, попавшей в образец[20]. ДНК животных плейстоцена Хорошо сохранившиеся образцы позволяют частично, а в некоторых случаях даже полностью секвенировать ядерный геном. В 2008 году была секвенирована ДНК, полученная из шерсти двух мамонтов, умерших около 20 и 59 тысяч лет назад. Картирование на черновую сборку генома африканского слона позволило оценить долю эндогенной ДНК в этих образцах в 90 % и 58 % соответственно; большую часть загрязнения в обоих случаях составили бактериальная ДНК и последовательности, происхождение которых определить не удалось. Полученные данные позволили оценить время расхождения между мамонтом и африканским слоном в 7,5 млн лет, а время расхождения между двумя линиями мамонта, для которых были взяты образцы, — в 1,5-2 млн лет. При этом секвенированная ДНК мамонтов совпадает со слоновьей на 99,41 % на нуклеотидном уровне и 99,78 % на аминокислотном уровне (то есть различие составляет примерно 1 остаток на белок). Принадлежность М4 и М25 к разным кладам была установлена ранее на основе секвенирования митохондриальной ДНК, и новая оценка на время расхождения согласуется с оценкой по мтДНК и уточняет последнюю. Одной из целей секвенирования мамонта было определение функционально важных аминокислотных различий между мамонтом и слоном. Были отобраны 92 различия между этими видами, которые могут иметь функциональное значение, и часть из которых, возможно, находилась под положительным отбором[21]. В одном из более ранних исследований в качестве образцов были взяты хорошо сохранившиеся при низких температурах останки восьми мамонтов из Музея мамонта в Хатанге. Для лучшего образца 45,4 % фрагментов ДНК выравнивались на сборку генома слона, при этом сходство между ДНК слона и мамонта составило 98,55 % без поправки на увеличение числа различий из-за дезаминации[22]. В 2013 году была получена черновая версия генома древней лошади[23]. Возраст образца оценивается в 560—780 тысяч лет. По состоянию на конец 2013 года это самый древний полный ядерный геном. Также были секвенированы ДНК лошади из позднего плейстоцена (~43 тысячи лет назад), пяти современных пород лошадей, лошади Пржевальского и осла. Филогенетический анализ показал, что последний общий предок всего рода Лошади жил 4-4,5 миллиона лет назад, что оказалось вдвое больше принятой до того оценки; популяции предков лошади Пржевальского и домашней лошади разошлись 38-72 тысячи лет назад. В том же году была восстановлена последовательность митохондриальной ДНК пещерного медведя из испанской Пещеры Костей, жившего в среднем плейстоцене, 179—680 тыс. лет назад, причём техника подготовки дДНК к секвенированию была оптимизирована для лучшего прочтения коротких (30-50 пн) фрагментов[10]. По состоянию на октябрь 2013-го, древний геном отсеквенирован с покрытием больше единицы только для таких позвоночных, как человек, белый медведь и лошадь. ДНК древних людей Неандертальцы На данный момент в европейской части Евразии найдено множество останков неандертальцев. Первым шагом в изучении их генетического материала была работа с митохондриальной ДНК, выделенной из костей, которые были обнаружены в 1856 году в долине реки Неандерталь (Германия). В 2006 году был начат проект по секвенированию полного генома неандертальца. В работе использовалась ДНК останков из пещеры Виндия в Хорватии, а также из некоторых других костей. Выравнивание полученного генома вместе с геномом современного человека и геномом шимпанзе, позволило грубо оценить время расхождения современного человека и неандертальца в 270—440 тыс. лет в предположении, что человек и шимпанзе разошлись 6,5 млн лет назад[24]. Геномы неандертальцев из пещеры Сидрон (англ.) (Испания), пещеры Фельдхофер (англ.) (Германия), Мезмайской пещеры (Россия) незначительно отличаются от первого. Секвенирование ДНК древних людей даёт надежду установить функциональные геномные мутации, которые отличают древнего человека от обезьяны, а также проследить отличия современного человека от древних людей. Исследование выявило удивительно малое количество зафиксировавшихся замен в геноме за довольно долгое время. Было найдено всего лишь 5 генов, в которых у современного человека зафиксировалось больше одной замены, меняющей структуру белка (по сравнению с неандертальцем, который в этих локусах совпал с шимпанзе): RPTN (англ.), SPAG17, CAN15, TTF1 (англ.) и PCD16. Сравнение разнообразия SNP для неандертальцев и современного человека позволяет локализовать мишени положительного отбора в геноме. Самый обширный такой участок генома, выявленный анализом различий SNP, принадлежит гену THADA (англ.), и SNP в ближайшем его окружении ассоциированы с диабетом 2 типа, а экспрессия этого гена существенно различается для больных диабетом и здоровых людей. В этом же регионе была обнаружена вставка 9 нуклеотидов в геноме человека, которая отсутствует у всех известных геномов от мыши до приматов и у неандертальца. Также SNP в других регионах ассоциируются с шизофренией, синдромом Дауна, аутизмом. Интерес представляет ген RUNX2 (англ.), который является единственным известным на данный момент геном, связанным с развитием ключично-черепного дизостоза. Анализ SNP также выявляет примесь генетического материала неандертальцев в ДНК неафриканцев, что подтверждает теорию о скрещивании неандертальцев с современными людьми, произошедшем после выхода популяции последних из Африки. Денисовцы Именно анализ древней ДНК позволил обнаружить этот вид древних людей — изначально считалось, что найденные в Денисовой пещере фрагменты скелета (две фаланги пальцев и три коренных зуба) принадлежат неандертальцам. Секвенирование митохондриальной ДНК останков опровергло теорию и показало, что они принадлежат отдельной группе. Различие денисовцев и современных людей в 2 раза превышает различие неандертальцев и современных людей, однако для ядерной ДНК эти различия имеют одинаковый порядок. Возможное объяснение состоит в том, что денисовцы и неандертальцы происходят от общего предка, который ранее отделился от ветви будущего современного человека. Эту гипотезу подтвердило сравнение двух геномов древних людей (денисовец, неандерталец) и пяти геномов современных людей (представителей Франции, Китая, Папуа — Новой Гвинеи, африканских народностей Йоруба и Бушменов), а также выравнивание геномов денисовцев, неандертальцев и африканцев Йоруба на геном шимпанзе. Помимо этого, исследование показало, что денисовцы и неандертальцы являются сестринскими группами. Также было показано, что денисовцы, как и неандертальцы, скрещивались с некоторыми неафриканскими популяциями современных людей: примесь ДНК неандертальцев присутствует во всех неафриканских группах, а примесь денисовцев — у папуасов и меланезийцев.[12]. Филогенетические выводы по данным ядерной и митохондриальной ДНК расходятся. Это может объясняться, тем, что мтДНК денисовцев происходит из некой древней линии, которая не прижилась в неандертальцах и современных людях. Большой размер древних популяций делает эту гипотезу правдоподобной, хотя для однозначного разрешения этой проблемы данных пока недостаточно. Гейдельбергский человек Анализ практически полного митохондриального генома гейдельбергского человека из Пещеры Костей позволил оценить возраст находки в 150—640 тысяч лет и показал, что митохондриальные геномы более схожи для гейдельбергского человека и денисовцев, чем для указанных видов и неандертальца, хотя по морфологическим признакам люди из Пещеры Костей близки к неандертальцам. Есть несколько возможных объяснений. Люди из Пещеры Костей могут представлять группу, отличную и от неандертальцев, и от денисовцев, которая внесла вклад в денисовскую мтДНК, но эта версия не объясняет морфологических черт неандертальцев у вида, напрямую с ними не связанного. Второе возможное объяснение состоит в том, что гейдельбергские люди относятся к общим предкам неандертальцев и денисовцев, однако эта версия предполагает существование в этой группе двух сильно разошедшихся линий мтДНК, предковых для неандертальцев и денисовцев. В третьих, не исключено влияние малоисследованных подвидов человека, которые могли сделать вклад в мтДНК гейдельберских людей и денисовцев. Анализ ядерной ДНК может прояснить картину[25]. Древняя эпигенетика Информация о метилировании цитозинов в CpG (англ.) может быть восстановлена непосредственно по данным секвенирования[26]. При дезаминировании, которое происходит случайно с течением времени, неметилированный цитозин превращается в урацил, а метилированный — в тимин. Протокол секвенирования древней ДНК предполагает обработку ДНК урацил-ДНК-гликозилазой и эндонуклеазой VIII, в результате которой урацил удаляется и молекула ДНК раскусывается в соответствующем месте с удалением поврежденного нуклеотида[27]. В результате при секвенировании для тех позиций, где цитозин был метилирован, будет получен большой процент чтений с Т, а неметилированный цитозин прочитается как C по тем молекулам, где дезаминирование в этом месте не произошло. По последовательности ДНК, извлечённой из волос 4000-летних останков палеоэскимоса удалось построить полногеномную карту расположения нуклеосом и метилирования[28], причём профиль метилирования оказался близок к тому, что наблюдается в волосах современного человека. Также восстановлена карта метилирования для неандертальцев и денисовцев[26][29]. Их метилом оказался похож на метилом современного человека, особенно в районе генов «домашнего хозяйства», однако найдено около 2000 областей со значимыми отличиями в метилировании. В частности, у древних людей найдены заметилированные участки в кластере HOXD, задействованном в регуляции развития конечностей, что может объяснять такие анатомические различия между ними и современными людьми, как менее длинные конечности, крупные кисти, широкие локтевые и коленные суставы[26][29]. Патогены Современные методы экстракции и секвенирования древней ДНК позволяют проводить исследования патогенов, полученных из останков давно умерших от болезни людей. Филогенетический анализ полученных образцов позволяет восстанавливать эволюцию болезнетворных организмов. Секвенированы средневековые штаммы чумной палочки и палочки Хансена, а также штамм палочки Коха XIX-го века. Чумная палочка Исследования дДНК чумной палочки (Yersinia pestis), полученных из лондонских захоронений жертв «Чёрной смерти» установили, что чумная палочка того времени, если не считать возможных геномных перестроек, характерных для этого вида, и ограничиться однонуклеотидными полиморфизмами, мало отличалась от современных штаммов. Более того, для всех полиморфизмов, отличающих её от современного штамма, в ней присутствовал вариант предка чумной палочки — Y. pseudotuberculosis. Эти данные позволяют предполагать, что генотип древней чумной палочки не являлся главной причиной её высокой вирулентности в то время и, возможно, его вклад был наравне с вкладом таких факторов, как генетически обусловленная восприимчивость носителей, климат, социальные условия, взаимодействия с другими болезнями. При помощи филогенетического анализа был установлен временной интервал, в который жил последний общий предок всех современных патогенных для человека штаммов чумной палочки: 1282—1343 года, причём древняя бактерия оказалась ближе всего к предковому узлу филогенетического дерева[30]. Фитофтора В 2013 году было опубликовано показательное исследование в области секвенирования старых патогенов, посвященное не существующим ныне штаммам фитофторы (Phytophthora infestans), которые вызвали Великий Картофельный Голод в Ирландии в середине XIX века. Генетический материал этих патогенов был выделен из сохраненных листьев картофеля и томата разного времени. До настоящего времени, ввиду отсутствия методов секвенирования, анализ данных был невозможен. Ученые провели анализ разных штаммов патогенов, выделив родственные штаммы из растений в Ирландии, Северной Америки и Европы. Исследования показали, что группа штаммов вызвавшая вышеупомянутый голод (HERB-1) и Северо-Американская группа (US-1) имели последнего общего предка предположительно на территории Мексики на рубеже XVIII и XIX веков. Это исследование особенно интересно с точки зрения подходов к анализу дДНК возбудителей прошедших эпидемий как животных, так и растений. Данная область находится на раннем этапе своего развития, однако в случае систематического создания биологических банков, подобные знания могут значительно способствовать ускоренному поиску противомикробных препаратов в ближайшем будущем, в случае возникновения эпидемий, вызванных родственными возбудителями[31]. См. такжеПримечания - ↑ 1 2 3 4 B. Shapiro, M. Hofreiter (24 January 2014). «A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA». Science 343 no. 6169: 3—16. DOI:10.1126/science.1236573.
- ↑ Higuchi R. et al. DNA sequences from the quagga, an extinct member of the horse family (англ.) // Nature. — 1985. — Vol. 312, no. 5991. — P. 282—284. — DOI:10.1038/312282a0. — PMID 6504142.
- ↑ Pääbo S. Molecular cloning of Ancient Egyptian mummy DNA (англ.) // Nature. — 1985. — Vol. 314, no. 6062. — P. 644—645. — DOI:10.1038/314644a0. — PMID 3990798.
- ↑ Pääbo S. Molecular genetic investigations of ancient human remains (англ.) // Cold Spring Harb Symp Quant Biol. — 1986. — Vol. 51, no. Pt 1. — P. 441—446. — PMID 3107879.
- ↑ 1 2 3 4 5 6 7 8 9 Eske Willerslev and Alan Cooper (January 2005). «Review Paper. Ancient DNA». Proc. R. Soc. B 272 no. 1558: 3—16. DOI:10.1098/rspb.2004.2813.
- ↑ Ermanno Rizzi, Martina Lari, Elena Gigli, Gianluca De Bellis, David Caramelli Ancient DNA studies: new perspectives on old samples (En) // Genetics Selection Evolution. — 2012/12. — Т. 44, вып. 1. — С. 21. — ISSN 1297-9686. — DOI:10.1186/1297-9686-44-21.
- ↑ Pontus Skoglund, Jan Storå, Anders Götherström, Mattias Jakobsson Accurate sex identification of ancient human remains using DNA shotgun sequencing // Journal of Archaeological Science. — Т. 40, вып. 12. — С. 4477–4482. — DOI:10.1016/j.jas.2013.07.004.
- ↑ Adrian M. Whatmore Ancient-Pathogen Genomics: Coming of Age? (англ.) // mBio. — 2014-10-31. — Vol. 5, iss. 5. — P. e01676–14. — ISSN 2150-7511. — DOI:10.1128/mbio.01676-14.
- ↑ 1 2 3 4 Michael Hofreiter, David Serre, Hendrik N. Poinar, Melanie Kuch and Svante Pääbo (2001). «Ancient DNA». Nature Reviews Genetics 2: 353—359. DOI:10.1038/35072071.
- ↑ 1 2 Jesse Dabney et al. (9 September 2013). «Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments». PNAS. DOI:10.1073/pnas.1314445110.
- ↑ Pontus Skoglund, Bernd H. Northoff, Michael V. Shunkov, Anatoli P. Derevianko, Svante Pääbo, Johannes Krause, and Mattias Jakobsson. (11 February 2014). «Separating endogenous ancient DNA from modern day contamination in a Siberian Neandertal». Proc. Natl. Acad. Sci. USA vol. 111 no. 6: 2229—2234. DOI:10.1073/pnas.1318934111. Этот метод требует наличия референсного генома. В первом приближении метод выглядит следующим образом. Чтения выравниваются на геном, после чего рассматриваются позиции, где встречаются, например, цитозин и тимин. Сравниваются две модели: одна предполагает, что замена цитозина на тимин произошла в результате посмертной деградации ДНК, вторая предполагает, что это полиморфизм или ошибка секвенирования. Модель выбирается по методу максимального правдоподобия.
- ↑ 1 2 David Reich et al. (23/30 December 2010). «Genetic history of an archaic hominin group from Denisova Cave in Siberia». Nature vol. 468: 1053—1060. DOI:10.1038/nature09710.
- ↑ Henrik B. Hansen, Peter B. Damgaard, Ashot Margaryan, Jesper Stenderup, Niels Lynnerup Comparing Ancient DNA Preservation in Petrous Bone and Tooth Cementum (англ.) // PLOS ONE. — 2017-01-27. — Vol. 12, iss. 1. — P. e0170940. — ISSN 1932-6203. — DOI:10.1371/journal.pone.0170940.
- ↑ Jesse Dabney, Michael Knapp, Isabelle Glocke, Marie-Theres Gansauge, Antje Weihmann Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments (англ.) // Proceedings of the National Academy of Sciences. — 2013-09-24. — Vol. 110, iss. 39. — P. 15758–15763. — ISSN 1091-6490 0027-8424, 1091-6490. — DOI:10.1073/pnas.1314445110.
- ↑ Iosif Lazaridis, Dani Nadel, Gary Rollefson, Deborah C. Merrett, Nadin Rohland Genomic insights into the origin of farming in the ancient Near East (En) // Nature. — 2016/08. — Т. 536, вып. 7617. — С. 419–424. — ISSN 1476-4687. — DOI:10.1038/nature19310.
- ↑ Beth Shapiro and Michael Hofreiter (eds.). Ancient DNA: Methods and Protocols. — Methods in Molecular Biology. — © Springer Science+Business Media, LLC 2012. — С. vol. 840. — ISBN DOI 10.1007/978-1-61779-516-9_10.
- ↑ Lindahl T. Instability and decay of the primary structure of DNA (англ.) // Nature. — 1993. — Vol. 362, no. 6422. — P. 709—715. — DOI:10.1038/362709a0. — PMID 8469282.
- ↑ S. Blair Hedges, Mary H. Schweitzer (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1191—1192. DOI:10.1126/science.7761839.
- ↑ В то время сборки человеческого генома ещё не существовало. См. Проект «Геном человека»
- ↑ Zischler H., Höss M., Handt O., von Haeseler A., van der Kuyl A.C., Goudsmit J. (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1193—4. DOI:10.1126/science.7605504.
- ↑ Web Miller et al. (20 november 2008). «Sequencing the nuclear genome of the extinct woolly mammoth». Nature vol. 456: 387—392. DOI:10.1038/nature07446.
- ↑ Hendrik N. Poinar et al. (20 January 2006). «Metagenomics to Paleogenomics: Large-Scale Sequencing of Mammoth DNA». Science vol. 311: 392—394. DOI:10.1126/science.1123360.
- ↑ Ludovic Orlando et al. (26 June 2013). «Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse». Nature vol. 499: 74—78. DOI:10.1038/nature12323.
- ↑ Richard E. Green et al. (7 May 2010). «A Draft Sequence of the Neandertal Genome». Science vol. 328: 710—722. DOI:10.1126/science.1188021.
- ↑ Matthias Meyer et al. (16 January 2014). «A mitochondrial genome sequence of a hominin from Sima de los Huesos». Nature 505: 403—406. DOI:10.1038/nature12788.
- ↑ 1 2 3 Elizabeth Pennisi (18 April 2014). «Ancient DNA Holds Clues to Gene Activity in Extinct Humans». Science Vol. 344 no. 6181: pp. 245-246. DOI:10.1126/science.344.6181.245.
- ↑ Briggs et al. (22 Dec 2009). «Removal of deaminated cytosines and detection of in vivo methylation in ancient DNA». Nucl. Acids Res. Vol. 38 (6): pp. e87. DOI:10.1093/nar/gkp1163.
- ↑ Jakob Skou Pedersen et al. (3 December 2013). «Genome-wide nucleosome map and cytosine methylation levels of an ancient human genome». Genome Res 24: 454—466. DOI:10.1101/gr.163592.113.
- ↑ 1 2 David Gokhman, Eitan Lavi, Kay Prüfer, Mario F. Fraga, José A. Riancho, Janet Kelso, Svante Pääbo, Eran Meshorer, Liran Carmel (April 17 2014). «Reconstructing the DNA Methylation Maps of the Neandertal and the Denisovan». Science Published Online. DOI:10.1126/science.1250368.
- ↑ Kirsten I. Bos, Verena J. Schuenemann, G. Brian Golding, Hernán A. Burbano, Nicholas Waglechner A draft genome of Yersinia pestis from victims of the Black Death (En) // Nature. — 2011/10. — Т. 478, вып. 7370. — С. 506–510. — ISSN 1476-4687. — DOI:10.1038/nature10549.
- ↑ Kentaro Yoshida Verena J Schuenemann Liliana M Cano Marina Pais Bagdevi Mishra Rahul Sharma Chirsta Lanz Frank N Martin Sophien Kamoun Johannes Krause Marco Thines Detlef Weigel Hernán A Burbano The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine (англ.) // eLife. — 2013. — Май.
Литератураhttp-wikipediya.ru Миф о древних ДНК-генеалогических линиях африканцев (часть 1) В академической литературе за последние четверть века регулярно появляются утверждения о том, что африканские ДНК-линии «самые древние», хотя основания для того не приводятся. И понятно, почему не приводятся – их просто нет. Давайте для начала разберемся, что такое «древние ДНК-линии», понимая под этим цепочки снипов Y-хромосомы.  Но разбираться особенно и не надо, потому что у всех мужчин на Земле, африканцев и неафриканцев, во всяком случае, у всех, кто делал тест на ДНК, снип-цепочки Y-хромосомы по древности одинаковы. У всех эти цепочки снипов тянутся от общих предков с шимпанзе, орангутангом, гориллой, макакой и другими древними общими с нами предками. Ниже – характерный пример, процитированный еще в 2012 году в журнале Advances in Anthropology (Klyosov, Rozhanskii, Ryabchenko): Но разбираться особенно и не надо, потому что у всех мужчин на Земле, африканцев и неафриканцев, во всяком случае, у всех, кто делал тест на ДНК, снип-цепочки Y-хромосомы по древности одинаковы. У всех эти цепочки снипов тянутся от общих предков с шимпанзе, орангутангом, гориллой, макакой и другими древними общими с нами предками. Ниже – характерный пример, процитированный еще в 2012 году в журнале Advances in Anthropology (Klyosov, Rozhanskii, Ryabchenko):  Сравнение фрагмента нуклеотидной последовательности Y-хромосомычеловека, шимпанзе, гориллы, орангутанга и макаки Видно, что в 90% случаев нуклеотидные последовательности показанных фагментов Y-хромосомы совпадают. А у шимпанзе и человека совпадают 93 нуклеотида из 97, то есть в 96%. Так что у всех нас ДНК-корни древние, и африканцы в этом отношении никак не выделяются. Хорошо, а насколько древние? Антропологи оценивают время расхождения линии будущего человека от линии будущего шимпанзе в 4-6 миллионов лет назад, это же повторяют и генетики, хотя поиски статей с конкретными расчетами результатов не дали. А вот результаты расчета с помощью автоматического калькулятора Килина-Клёсова (ККК, 2015), иллюстрация увеличивается по клику: 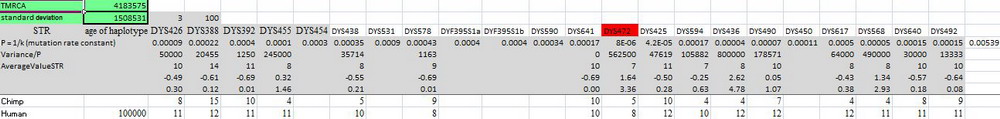 Общий предок человека и шимпанзе жил, если округлить расчеты, 4.2±1.5 миллионов лет назад. 16-маркерный гаплотип шимпанзе был извлечен из геномной базы данных, как описано в той статье в Advances in Anthropology (2012), а в качестве обобщенного гаплотипа человека брался экстраполированный предковый гаплотип по всем неафриканским гаплогруппам, пересчитанный на времена 100 тысяч лет назад. Впрочем, можно было и не пересчитывать, вот – время до общего предка современных носителей гаплогруппы А00 и шимпанзе, иллюстрация увеличивается по клику: Общий предок человека и шимпанзе жил, если округлить расчеты, 4.2±1.5 миллионов лет назад. 16-маркерный гаплотип шимпанзе был извлечен из геномной базы данных, как описано в той статье в Advances in Anthropology (2012), а в качестве обобщенного гаплотипа человека брался экстраполированный предковый гаплотип по всем неафриканским гаплогруппам, пересчитанный на времена 100 тысяч лет назад. Впрочем, можно было и не пересчитывать, вот – время до общего предка современных носителей гаплогруппы А00 и шимпанзе, иллюстрация увеличивается по клику:  Оно равно 4.4±1.4 миллионов лет назад, что практически одинаково в пределах погрешности расчетов. Те же времена дают все основные гаплогруппы серии А, например: • А0а-V150 имеет общего предка с шимпанзе 4.6±1.7 млн лет назад; • A1b1-M32 (Чад и Турция) 4.3±1.4 млн лет назад; • A1b1-M32, M13 (Британия) 4.2±1.4 млн лет назад, а также • гаплогруппа В, 4.4±1.4 млн лет назад. Короче, в отношении «древних ДНК-линий» африканцы ничем от неафриканцев не отличаются. И те, и другие тянут свои ДНК-линии от общего предка с шимпанзе. И у тех, и у других цепочки снипов уходят на глубины в сотни тысяч и миллионы лет. К этому мы еще вернемся. Может, под «древними африканскими ДНК-линиями» попгенетики и прочие генетики подразумевают древние времена жизни их общих предков, и полагают, что они намного более древние, чем у неафриканцев? Придется их огорчить, ситуация противоположная. Африканские предки группы А (популяций, субкладов) почти все совсем недавние. Возьмем, например, гаплогруппу А00, которая среди гаплогрупп считается самой древней. Да, для нее цепочка снипов насчитывает – по данным, приводимым YFull – 1684 снипов, что при 144 годах на снип (это число приводит YFull) дает 242500 лет от начала гаплогруппы. Действительно, YFull приводят близкий «возраст» А00, 235 тысяч лет. Но откуда эта цепочка снипов ведет – никто не знает. Может, из Африки, может, с Кавказа, может, из Сибири, может, из Европы – неизвестно. Почему-то считается, что именно из Африки. Но это опять «по понятиям», правда, откуда появившимся, непонятно, извините за игру слов. Древнейшие ДНК находили в Европе, на Русской равнине, в Сибири, а в Африке ни разу, правда, и исследований таких не было. Так откуда эти «понятия» появились? А так, стиль попгенетиков. Кстати, о возрасте гаплогруппы А00. Калькулятор КК дает для базовых гаплотипов А0 и А00 возраст общего предка (с округлением) 217000±87000 лет. Это – возраст А00 или немного старше. Так что датировка по снипам воспроизводится по гаплотипам в пределах погрешности расчетов (иллюстрация ниже увеличивается по клику). Оно равно 4.4±1.4 миллионов лет назад, что практически одинаково в пределах погрешности расчетов. Те же времена дают все основные гаплогруппы серии А, например: • А0а-V150 имеет общего предка с шимпанзе 4.6±1.7 млн лет назад; • A1b1-M32 (Чад и Турция) 4.3±1.4 млн лет назад; • A1b1-M32, M13 (Британия) 4.2±1.4 млн лет назад, а также • гаплогруппа В, 4.4±1.4 млн лет назад. Короче, в отношении «древних ДНК-линий» африканцы ничем от неафриканцев не отличаются. И те, и другие тянут свои ДНК-линии от общего предка с шимпанзе. И у тех, и у других цепочки снипов уходят на глубины в сотни тысяч и миллионы лет. К этому мы еще вернемся. Может, под «древними африканскими ДНК-линиями» попгенетики и прочие генетики подразумевают древние времена жизни их общих предков, и полагают, что они намного более древние, чем у неафриканцев? Придется их огорчить, ситуация противоположная. Африканские предки группы А (популяций, субкладов) почти все совсем недавние. Возьмем, например, гаплогруппу А00, которая среди гаплогрупп считается самой древней. Да, для нее цепочка снипов насчитывает – по данным, приводимым YFull – 1684 снипов, что при 144 годах на снип (это число приводит YFull) дает 242500 лет от начала гаплогруппы. Действительно, YFull приводят близкий «возраст» А00, 235 тысяч лет. Но откуда эта цепочка снипов ведет – никто не знает. Может, из Африки, может, с Кавказа, может, из Сибири, может, из Европы – неизвестно. Почему-то считается, что именно из Африки. Но это опять «по понятиям», правда, откуда появившимся, непонятно, извините за игру слов. Древнейшие ДНК находили в Европе, на Русской равнине, в Сибири, а в Африке ни разу, правда, и исследований таких не было. Так откуда эти «понятия» появились? А так, стиль попгенетиков. Кстати, о возрасте гаплогруппы А00. Калькулятор КК дает для базовых гаплотипов А0 и А00 возраст общего предка (с округлением) 217000±87000 лет. Это – возраст А00 или немного старше. Так что датировка по снипам воспроизводится по гаплотипам в пределах погрешности расчетов (иллюстрация ниже увеличивается по клику).  Так вот, об общих предках африканских популяций, субкладов группы А. Они почти все по времени очень мелкие, часто всего несколько сотен лет. Иногда – между 1000 и 2000 лет. Самые древние – примерно 5000-7000 лет. И опять – где эти предки жили, никто не знает. Взглянем на гаплогруппу А00. В литературе (Mendez et al., 2013) описаны 11 гаплотипов в 95-маркерном формате (без 16 мультимаркерных гаплотипов) носителей гаплогруппы А00, все из племени Mbo из Камеруна, и еще один – афроамериканец. Расчетов в статье, конечно, нет. Все 11 гаплотипов содержат 29 мутаций, что дает 29/11/0.161 = 16 условных поколений, но с учетом того, что 45 аллелей (из 1045 аллелей) авторы определить не смогли, то до общего предка на самом деле 17 условных поколений, то есть 425±90 лет. Итак, сама гаплогруппа А00 образовалась более 200 тысяч лет назад, причем неизвестно где, а общий предок носителей А00 из Камеруна жил примерно 425 лет назад. Если добавить к серии из Камеруна еще гаплотип афроамериканца той же гаплогруппы, который отличается от предкового гаплотипа камерунцев на 11 мутаций, то есть на 11/0.161 = 68 → 73 условных поколения, то есть на 1825 лет, то их общий предок, камерунцев и афроамериканца, жил примерно (425+1825)/2 = 1125 лет назад. Примеров недавних общих предков разных мужских популяций гаплогрупп класса А, которые сейчас живут в Африке, можно привести немало. В большинстве этих популяций гаплотипы или одинаковы или почти одинаковы, даже для протяженных гаплотипов. С доступными гаплотипами гаплогруппы А00 мы уже разобрались, им четыре сотни лет. Взглянем теперь на Проект FTDNA гаплогруппы А: • В гаплогруппе/субкладе А0-V150 имеются два 25-маркерных гаплотипа, оба идентичны друг другу, то есть их общий предок жил не более 300-400 лет назад. Других гаплотипов в 25-маркерном формате или выше нет. • В гаплогруппе A0a-L981 (L994, V148, V166) между двумя доступными 67-маркерными гаплотипами есть 8 мутаций, что помещает их общего предка на 900 лет назад. • В гаплогруппе A0b-V166, L896 шесть 37-маркерных гаплотипов имеют общего предка 475±160 лет назад. • В гаплогруппе A1a-M31 («европейский кластер») пять 37-маркерных гаплотипов имеют общего предка 1175±290 лет назад. • В гаплогруппе A1a-M31 (все неизвестного происхождения) пять 37-маркерных гаплотипов имеют общего предка 1880±390 лет назад. • В гаплогруппе A1a-M31 (из 18 гаплотипов – 14 неизвестного происхождения, два из Англии, один из Швейцарии, один Кейп Верде) все 37-маркерные, все имеют общего предка 730±130 лет назад. • В гаплогруппе A1a-M31 («британский кластер») 19 гаплотипов в 67-маркерном формате имеют общего предка 740±120 лет назад. В итоге, 50 гаплотипов в 37-маркерном формате четырех предшествующих серий (А0а-М31) имеют общего предка 9270±970 лет назад. Но надо обратить внимание, что это четыре серии гаплотипов, разбросанные по разным континентам. Это – не одна «ДНК-линия», это – четыре разных ДНК-линии, с четырьмя общими предками, каждый «глубиной» от 700 до 1900 лет. Ну и где, позволительно спросить, «древнейшие ДНК-линии в Африке»? Это – миф, ничем не обоснованный. Ископаемых ДНК нет, древних линий, как мы видим, нет, к тому же, в Африке вовсе не гаплотипы группы А явяются доминирующими. Доминирует гаплогруппа E1b; в Камеруне и Чаде, и отчасти в Нигерии, доминирует гаплогруппа R1b, поэтому неудивительно, что в Африке наблюдается «высокое генетическое разнообразие», только это не от древности, а от обилия носителей разных гаплогрупп, прибывших в Африку в разные времена. Да и вообще, как можно серьезно обращать внимание на «генетическое разнобразие» в открытых системах, какой является Африка? Как я уже не раз отмечал, самым «генетически разнообразным» городом мира является, наверное, Нью-Йорк, но вряд ли кто будет серьезно утверждать, что Нью-Йорк – древнейший город мира. Кроме, наверное, популяционных генетиков, с их примитивными и бездумными критериями. Откуда же появился этот миф о «древности африканских линий»? Датировок ведь их до последнего времени не было, да и датировки бывают, как мы убедились, нескольких видов – цепочек мутаций, уходящих далеко вглубь, до приматов; цепочек мутаций, ведущих ко временам образования соответствующих гаплогрупп; и датировок общих предков современных носителей соответствующих гаплогрупп и субкладов. Ни одна из этих датировок не «укоренена» территориально, то есть мы не знаем, на какой территории, на каком континенте образовалась та или иная гаплогруппа. Мы можем только набирать сведения и складывать фрагменты общей картины, но Африка в этом отношении находится на одном из последних мест по накопленным знаниям. Но фантазий, натяжек и откровенных фальсификаций про них со стороны попгенетиков – хоть отбавляй. Собственно, это характерно для популяционных генетиков, но они уроки из принципиальных ошибок не извлекают. Еще не так давно они утверждали, что гаплогруппа R1b образовалась в Европе 30 тысяч лет назад, и сначала приняли в штыки, а потом напрочь игнорировали сведения и датировки ДНК-генеалогии, что R1b прибыла из Южной Сибири длинным миграционным путем в Европу примерно 4800 лет назад (Klyosov, 2009; Klyosov, 2012). Называли это «псевдонаукой», особенно в этом усердствовали попгенетики Балановские. Разворот произошел в 2013 году, когда неподалеку от Байкала нашли ископаемую гаплогруппу R с археологической датировкой 24 тысячи лет назад. Лед тронулся, и попгенетики, не моргнув глазом, не повинившись и не цитируя статьи с датировками ДНК-генеалогии, стали повторять то, что уже было опубликовано нами несколько лет назад. Например, в недавней статье Batini, Jobling и других (2015) датировки гаплогруппы R1b в Европе уже даются как R1b-L11 4510 лет (доверительный интервал 3920-5160 лет назад) и R1b-P312 4210 лет (доверительный интервал 3700-4790 лет назад). YFull дают датировки R1b-U106 4900 лет (доверительный интервал 4400-5400 лет назад), R1b-P312 4600 лет (для TMRCA, доверительный интервал 4300-5000 лет назад). К сожалению, приходится повторять, что попгенетики уроков из своих ошибок не извлекают. Ситуация повторяется с Африкой. Данных нет, или данные говорят о другом, но их придумывают и искажают. Зачем это им нужно? Для нормального человека звучит невероятно, но это ментальность вассалов, которые лебезят перед сюзереном. Сюзерен решает, кого пригреть, кого пригласить на конференцию, кому дать грант, кого взять в коллективную статью, ввести в комиссию или комитет, в общем, в инфраструктуру. Без сюзерена вассал – никто, и вассал это прекрасно понимает. А зачем это нужно сюзерену? О, там большая политика. Вассала к этим вопросам не допускают. Вассал знает, что если он публично усомнится в африканском происхождении человечества, то его дни сочтены. Поэтому он, вассал, готов разорвать на части любого, кто сомневается или выставляет данные против. А поскольку он разорвать на части не может, то цель – найти компромат, состряпать ложь, передернуть, исказить – других способов или аргументов у вассала нет, во всяком случае, научных. Посмотрим на схему гаплогрупп и субкладов группы А, приведенную согласно классификации ISOGG, в сокращенном виде: Так вот, об общих предках африканских популяций, субкладов группы А. Они почти все по времени очень мелкие, часто всего несколько сотен лет. Иногда – между 1000 и 2000 лет. Самые древние – примерно 5000-7000 лет. И опять – где эти предки жили, никто не знает. Взглянем на гаплогруппу А00. В литературе (Mendez et al., 2013) описаны 11 гаплотипов в 95-маркерном формате (без 16 мультимаркерных гаплотипов) носителей гаплогруппы А00, все из племени Mbo из Камеруна, и еще один – афроамериканец. Расчетов в статье, конечно, нет. Все 11 гаплотипов содержат 29 мутаций, что дает 29/11/0.161 = 16 условных поколений, но с учетом того, что 45 аллелей (из 1045 аллелей) авторы определить не смогли, то до общего предка на самом деле 17 условных поколений, то есть 425±90 лет. Итак, сама гаплогруппа А00 образовалась более 200 тысяч лет назад, причем неизвестно где, а общий предок носителей А00 из Камеруна жил примерно 425 лет назад. Если добавить к серии из Камеруна еще гаплотип афроамериканца той же гаплогруппы, который отличается от предкового гаплотипа камерунцев на 11 мутаций, то есть на 11/0.161 = 68 → 73 условных поколения, то есть на 1825 лет, то их общий предок, камерунцев и афроамериканца, жил примерно (425+1825)/2 = 1125 лет назад. Примеров недавних общих предков разных мужских популяций гаплогрупп класса А, которые сейчас живут в Африке, можно привести немало. В большинстве этих популяций гаплотипы или одинаковы или почти одинаковы, даже для протяженных гаплотипов. С доступными гаплотипами гаплогруппы А00 мы уже разобрались, им четыре сотни лет. Взглянем теперь на Проект FTDNA гаплогруппы А: • В гаплогруппе/субкладе А0-V150 имеются два 25-маркерных гаплотипа, оба идентичны друг другу, то есть их общий предок жил не более 300-400 лет назад. Других гаплотипов в 25-маркерном формате или выше нет. • В гаплогруппе A0a-L981 (L994, V148, V166) между двумя доступными 67-маркерными гаплотипами есть 8 мутаций, что помещает их общего предка на 900 лет назад. • В гаплогруппе A0b-V166, L896 шесть 37-маркерных гаплотипов имеют общего предка 475±160 лет назад. • В гаплогруппе A1a-M31 («европейский кластер») пять 37-маркерных гаплотипов имеют общего предка 1175±290 лет назад. • В гаплогруппе A1a-M31 (все неизвестного происхождения) пять 37-маркерных гаплотипов имеют общего предка 1880±390 лет назад. • В гаплогруппе A1a-M31 (из 18 гаплотипов – 14 неизвестного происхождения, два из Англии, один из Швейцарии, один Кейп Верде) все 37-маркерные, все имеют общего предка 730±130 лет назад. • В гаплогруппе A1a-M31 («британский кластер») 19 гаплотипов в 67-маркерном формате имеют общего предка 740±120 лет назад. В итоге, 50 гаплотипов в 37-маркерном формате четырех предшествующих серий (А0а-М31) имеют общего предка 9270±970 лет назад. Но надо обратить внимание, что это четыре серии гаплотипов, разбросанные по разным континентам. Это – не одна «ДНК-линия», это – четыре разных ДНК-линии, с четырьмя общими предками, каждый «глубиной» от 700 до 1900 лет. Ну и где, позволительно спросить, «древнейшие ДНК-линии в Африке»? Это – миф, ничем не обоснованный. Ископаемых ДНК нет, древних линий, как мы видим, нет, к тому же, в Африке вовсе не гаплотипы группы А явяются доминирующими. Доминирует гаплогруппа E1b; в Камеруне и Чаде, и отчасти в Нигерии, доминирует гаплогруппа R1b, поэтому неудивительно, что в Африке наблюдается «высокое генетическое разнообразие», только это не от древности, а от обилия носителей разных гаплогрупп, прибывших в Африку в разные времена. Да и вообще, как можно серьезно обращать внимание на «генетическое разнобразие» в открытых системах, какой является Африка? Как я уже не раз отмечал, самым «генетически разнообразным» городом мира является, наверное, Нью-Йорк, но вряд ли кто будет серьезно утверждать, что Нью-Йорк – древнейший город мира. Кроме, наверное, популяционных генетиков, с их примитивными и бездумными критериями. Откуда же появился этот миф о «древности африканских линий»? Датировок ведь их до последнего времени не было, да и датировки бывают, как мы убедились, нескольких видов – цепочек мутаций, уходящих далеко вглубь, до приматов; цепочек мутаций, ведущих ко временам образования соответствующих гаплогрупп; и датировок общих предков современных носителей соответствующих гаплогрупп и субкладов. Ни одна из этих датировок не «укоренена» территориально, то есть мы не знаем, на какой территории, на каком континенте образовалась та или иная гаплогруппа. Мы можем только набирать сведения и складывать фрагменты общей картины, но Африка в этом отношении находится на одном из последних мест по накопленным знаниям. Но фантазий, натяжек и откровенных фальсификаций про них со стороны попгенетиков – хоть отбавляй. Собственно, это характерно для популяционных генетиков, но они уроки из принципиальных ошибок не извлекают. Еще не так давно они утверждали, что гаплогруппа R1b образовалась в Европе 30 тысяч лет назад, и сначала приняли в штыки, а потом напрочь игнорировали сведения и датировки ДНК-генеалогии, что R1b прибыла из Южной Сибири длинным миграционным путем в Европу примерно 4800 лет назад (Klyosov, 2009; Klyosov, 2012). Называли это «псевдонаукой», особенно в этом усердствовали попгенетики Балановские. Разворот произошел в 2013 году, когда неподалеку от Байкала нашли ископаемую гаплогруппу R с археологической датировкой 24 тысячи лет назад. Лед тронулся, и попгенетики, не моргнув глазом, не повинившись и не цитируя статьи с датировками ДНК-генеалогии, стали повторять то, что уже было опубликовано нами несколько лет назад. Например, в недавней статье Batini, Jobling и других (2015) датировки гаплогруппы R1b в Европе уже даются как R1b-L11 4510 лет (доверительный интервал 3920-5160 лет назад) и R1b-P312 4210 лет (доверительный интервал 3700-4790 лет назад). YFull дают датировки R1b-U106 4900 лет (доверительный интервал 4400-5400 лет назад), R1b-P312 4600 лет (для TMRCA, доверительный интервал 4300-5000 лет назад). К сожалению, приходится повторять, что попгенетики уроков из своих ошибок не извлекают. Ситуация повторяется с Африкой. Данных нет, или данные говорят о другом, но их придумывают и искажают. Зачем это им нужно? Для нормального человека звучит невероятно, но это ментальность вассалов, которые лебезят перед сюзереном. Сюзерен решает, кого пригреть, кого пригласить на конференцию, кому дать грант, кого взять в коллективную статью, ввести в комиссию или комитет, в общем, в инфраструктуру. Без сюзерена вассал – никто, и вассал это прекрасно понимает. А зачем это нужно сюзерену? О, там большая политика. Вассала к этим вопросам не допускают. Вассал знает, что если он публично усомнится в африканском происхождении человечества, то его дни сочтены. Поэтому он, вассал, готов разорвать на части любого, кто сомневается или выставляет данные против. А поскольку он разорвать на части не может, то цель – найти компромат, состряпать ложь, передернуть, исказить – других способов или аргументов у вассала нет, во всяком случае, научных. Посмотрим на схему гаплогрупп и субкладов группы А, приведенную согласно классификации ISOGG, в сокращенном виде:  В этой схеме – источник принципиальной путаницы попенетиков. Они не смогли увидеть и осознать, что эта лесенка субкладов представляет собой серию вилок, которые периодически расходятся на «африканские» (по сегодняшнему месту жительства) и неафриканские ветви. Опишем кратко эти вилки, а потом рассмотрим их подробнее – когда они образовались, и кто являются потомками субкладов по обе стороны тех вилок. Итак, вилок всего четыре. Первой от ствола, уходящего вглубь эволюционного дерева человечества (точнее, его Y-хромосомы), отходит вилка, знаменующая, видимо, начало Homo sapiens – расхождение на А00 и А0-Т. Вторая – расхождение А0-Т на А0 и А1. Третья – расхождение А1 на А1a и A1b. Четвертая – расхождение A1b на A1b1 и ВТ. Если упрощенно – то каждая вилка расходится на африканскую (по нынешнему месту жительства) и неафриканскую стороны. А00, А0, A1a и A1b1 уходят в Африку, противоположные стороны вилки A0-T, A1, A1b и BT остаются вне Африки. «Упрощенно» – потому что вилка на А00 и A0-T образовалась еще до расхождения на африканцев и неафриканцев (это расхождение произошло примерно 160 тысяч лет назад), а впоследствии многие неафриканцы перебирались в Африку – это и многочисленные носители гаплогрупп E1b, J2, R1b, и так далее. Среди них – миллионы современных жителей Западной Африки (берберы, жители Камеруна и Чада), египтяне, потомки буров в Южной Африке, и так далее. Немало жителей Европы и Ближнего Востока имеют гаплогруппу A1a, как недавно показывал И.Л. Рожанский в статье на Переформате. Да и вообще носителей группы А в Африке относительно мало – часто на уровне единиц процента или того меньше. Рассмотрим вилки подробнее. Первая вилка – расхождение предшественника Homo sapiens (или Homo sapiens в начальной стадии, примерно 230 тысяч лет назад) на гаплогруппы А00 и А0-Т. Нисходящих субкладов от А00 пока не обнаружено, и ее нынешние носители обнаружены только в Африке в количестве 11 человек (см. выше), с общим предком несколько сотен лет назад, а также несколько афроамериканцев с одинаковыми гаплотипами. Снипы гаплогруппы А0-Т, напротив, имеются у всех неафриканцев, в том числе у всех нас, читателей Переформата, хотя только у мужчин. И, разумеется, у всех африканцев, носителей гаплогрупп E1b, J2, R1b, В и других, которые прибыли в Африку относительно недавно. Но помимо них снипы группы А0-Т должны быть и у всех африканцев, носителей групп А, потому эти снипы предшествуют разделению африканских и неафриканских линий (примерно 160 тысяч лет назад). Впрочем, речь идет о промежуточных снипах в цепочках наших и африканских снипов, носителей А0-Т как терминальных снипов пока ни у кого не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» никакого смысла не имеет. К тому, что у всех нас, мужчин, есть множество снипов гаплогруппы А0-Т – ниже приведен список части из 587 снипов этой гаплогруппы, из которых у меня есть L1090, L1121, L1124, L1125, L1127, L1235 и многие другие: Мой список снипов BigY CTS10042, CTS10627, CTS10834, CTS10847, CTS10976, CTS10993, CTS11148, CTS11184, CTS11411, CTS11468, CTS11530, CTS1169, CTS11720, CTS11734, CTS11853, CTS11913, CTS11948, CTS12010, CTS1211, CTS1277, CTS1278, CTS12947, CTS12972, CTS1340, CTS1367, CTS1415, CTS1416, CTS1417, CTS1419, CTS1422, CTS1619, CTS1738, CTS1795, CTS1907, CTS2134, CTS262, CTS2626, CTS2636, CTS2637, CTS2638, CTS2639, CTS2663, CTS 2680, CTS2891, CTS2988, CTS2992, CTS3067, CTS3075, CTS3229, CTS3230, CTS3231, CTS3316, CTS3321, CTS3358, CTS3402, CTS3441, CTS3527, CTS3534, CTS3548, CTS3551, CTS358, CTS3607, CTS3622, CTS3649, CTS3650, CTS3654, CTS3697, CTS3775, CTS3848, CTS3943, CTS3984, CTS4259, CTS4465, CTS4862, CTS501, CTS5287, CTS5423, CTS5454, CTS5508, CTS5577, CTS5648, CTS570, CTS5936, CTS5979, CTS623, CTS6918, CTS6985, CTS7191, CTS7275, CTS7278, CTS7400, CTS7500, CTS7650, CTS7659, CTS7941, CTS8008, CTS8127, CTS8350, CTS8507, CTS8508, CTS8591, CTS8626, CTS8665, CTS8797, CTS8816, CTS8896, CTS9018, CTS903, CTS9108, CTS9510, CTS9515, CTS9539, CTS9596, CTS9733, CTS9739, CTS9754, CTS9779, F1050, F1209, F1224, F132, F1586, F184, F185, F2957, F3032, F313, F3159, F3166, F3185, F3194, F3197, F3337, F3561, F3564, F3644, F3650, F3692, F4099, F686, F719, F773, F82, F937, F989, IMS-JST002612, IMS-ST022448_3, IMS-ST022448_6, IMS-ST022449_3, IMS-ST022449_6, IMS-ST022451_2, K511, L1002, L1004, L1005, L1009, L1013, L1028, L104, L105, L1053, L1060, L1061, L1062, L1067, L1071, L108, L1089, L1090, L1095, L1098, L1099, L110, L1101, L1105, L1112, L1116, L1118, L112, L1120, L1121, L1123, L1124, L1125, L1127, L1129, L113, L1132, L1135, L1136, L1137, L114, L1142, L1143, L1145, L1150, L1155, L1179, L1185, L1120, L1225, L1235, L132, L1346, L1347, L1352, L1480, L1492, L15, L16, L168, L248, L268, L313, L350, L352, L413, L418, L438, L440, L449, L457, L468, L469, L498, L543, L58, L604, L721, L74, L741, L747, L76, L760, L768, L777, L779, L781, L819, L82, L821, L851, L875, L882, L962, L969, L970, L977, L986, L989, M168, M173, M198, M207, M213, M235, M251, M294, M299, M306, M417, M42, M420, M448, M449, M45, M459, M511, M512, M513, M514, M515, M516, M526, M74, M89, M9, M94, P108, P128, P131, P132, P133, P134, P135, P136, P138, P139, P14, P140, P141, P143, P145, P146, P148, P149, P151, P157, P158, P159, P160, P161, P163, P166, P187, P19_1, P207, P224, P225, P226, P228, P229, P230, P231, P232, P233, P234, P235, P236, P237, P238, P239, P242, P243, P244, P245, P27.1_2, P280, P281, P282, P283, P284, P285, P286, P294, P305, P316, PAGES00026, PAGES00081, PAGES00083, PF1345, PF1378, PF1389, PF1396, PF1402, PF1403, PF1405, PF1407, PF1408, PF1411, PF1412, PF1414, PF1415, PF1418, PF1419, PF2589, PF2590, PF2591, PF2608, PF2610, PF2611, PF2614, PF2615, PF2616, PF2619, PF2620, PF2621, PF2622, PF2626, PF2629, PF2631, PF2634, PF2635, PF2637, PF2640, PF2642, PF2647, PF2651, PF2653, PF2658, PF2660, PF2683, PF2685, PF288, PF2690, PF2694, PF2695, PF2700, PF2709, PF2716, PF2718, PF2722, PF2723, PF2729, PF2731, PF2732, PF2736, PF2737, PF2739, PF2742, PF2747, PF2748, PF2749, PF2752, PF2753, PF2754, PF2755, PF2756, PF2757, PF2758, PF2770, PF2772, PF2988, PF349, PF3494, PF3495, PF3496, PF3497, PF3500, PF4522, PF5459, PF5469, PF5477, PF5483, PF5488, PF5490, PF5498, PF5500, PF5503, PF5513, PF5861, PF5862, PF5863, PF5864, PF5869, PF5870, PF5871, PF5872, PF5876, PF5881, PF5882, PF5885, PF5886, PF5888, PF5889, PF5895, PF5908, PF5911, PF5914, PF5916, PF5919, PF5920, PF5936, PF5940, PF5943, PF5944, PF5945, PF5949, PF5951, PF5953, PF5954, PF5955, PF5956, PF5957, PF5958, PF5964, PF5965, PF5967, PF5970, PF5971, PF5975, PF5978, PF5980, PF5981, PF5982, PF5984, PF5985, PF5991, PF6013, PF6016, PF6047, PF6054, PF6056, PF6057, PF6066, PF6069, PF6071, PF6072, PF6073, PF6114, PF6116, PF6118, PF6121, PF6132, PF6133, PF6143, PF6153, PF6156, PF6158, PF6159, PF6160, PF6162, PF6163, PF6165, PF6168, PF6169, PF6170, PF6172, PF6179, PF6181, PF6194, PF6195, PF6201, PF6204, PF6211, PF6212, PF6213, PF6214, PF6215, PF6216, PF6217, PF6218, PF6220, PF6222, PF6230, PF6231, PF6233, PF6512, PF682, PF7514, PF7522, PF7527, PF7530, PF7531, PF7532, PF7534, PF7540, PF7542, PK1, PR1220, PR202, PR2119, PR5761, PR855, rs161492_1, V221, V238, V241, V250, Y2902, Y32, Y33, Y34, Y36, Y783.2, YSC0000057, YSC0000077, YSC0000081, YSC0000107, YSC0000108, YSC0000109, YSC0000110, YSC0000193, YSC0000350, YSC0000913, Z1331, Z280, Z282, Z5997, Z6030, Z6032. Фрагмент из списка снипов гаплогруппы A0-T (приведены 182 снипа из 587) В этой схеме – источник принципиальной путаницы попенетиков. Они не смогли увидеть и осознать, что эта лесенка субкладов представляет собой серию вилок, которые периодически расходятся на «африканские» (по сегодняшнему месту жительства) и неафриканские ветви. Опишем кратко эти вилки, а потом рассмотрим их подробнее – когда они образовались, и кто являются потомками субкладов по обе стороны тех вилок. Итак, вилок всего четыре. Первой от ствола, уходящего вглубь эволюционного дерева человечества (точнее, его Y-хромосомы), отходит вилка, знаменующая, видимо, начало Homo sapiens – расхождение на А00 и А0-Т. Вторая – расхождение А0-Т на А0 и А1. Третья – расхождение А1 на А1a и A1b. Четвертая – расхождение A1b на A1b1 и ВТ. Если упрощенно – то каждая вилка расходится на африканскую (по нынешнему месту жительства) и неафриканскую стороны. А00, А0, A1a и A1b1 уходят в Африку, противоположные стороны вилки A0-T, A1, A1b и BT остаются вне Африки. «Упрощенно» – потому что вилка на А00 и A0-T образовалась еще до расхождения на африканцев и неафриканцев (это расхождение произошло примерно 160 тысяч лет назад), а впоследствии многие неафриканцы перебирались в Африку – это и многочисленные носители гаплогрупп E1b, J2, R1b, и так далее. Среди них – миллионы современных жителей Западной Африки (берберы, жители Камеруна и Чада), египтяне, потомки буров в Южной Африке, и так далее. Немало жителей Европы и Ближнего Востока имеют гаплогруппу A1a, как недавно показывал И.Л. Рожанский в статье на Переформате. Да и вообще носителей группы А в Африке относительно мало – часто на уровне единиц процента или того меньше. Рассмотрим вилки подробнее. Первая вилка – расхождение предшественника Homo sapiens (или Homo sapiens в начальной стадии, примерно 230 тысяч лет назад) на гаплогруппы А00 и А0-Т. Нисходящих субкладов от А00 пока не обнаружено, и ее нынешние носители обнаружены только в Африке в количестве 11 человек (см. выше), с общим предком несколько сотен лет назад, а также несколько афроамериканцев с одинаковыми гаплотипами. Снипы гаплогруппы А0-Т, напротив, имеются у всех неафриканцев, в том числе у всех нас, читателей Переформата, хотя только у мужчин. И, разумеется, у всех африканцев, носителей гаплогрупп E1b, J2, R1b, В и других, которые прибыли в Африку относительно недавно. Но помимо них снипы группы А0-Т должны быть и у всех африканцев, носителей групп А, потому эти снипы предшествуют разделению африканских и неафриканских линий (примерно 160 тысяч лет назад). Впрочем, речь идет о промежуточных снипах в цепочках наших и африканских снипов, носителей А0-Т как терминальных снипов пока ни у кого не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» никакого смысла не имеет. К тому, что у всех нас, мужчин, есть множество снипов гаплогруппы А0-Т – ниже приведен список части из 587 снипов этой гаплогруппы, из которых у меня есть L1090, L1121, L1124, L1125, L1127, L1235 и многие другие: Мой список снипов BigY CTS10042, CTS10627, CTS10834, CTS10847, CTS10976, CTS10993, CTS11148, CTS11184, CTS11411, CTS11468, CTS11530, CTS1169, CTS11720, CTS11734, CTS11853, CTS11913, CTS11948, CTS12010, CTS1211, CTS1277, CTS1278, CTS12947, CTS12972, CTS1340, CTS1367, CTS1415, CTS1416, CTS1417, CTS1419, CTS1422, CTS1619, CTS1738, CTS1795, CTS1907, CTS2134, CTS262, CTS2626, CTS2636, CTS2637, CTS2638, CTS2639, CTS2663, CTS 2680, CTS2891, CTS2988, CTS2992, CTS3067, CTS3075, CTS3229, CTS3230, CTS3231, CTS3316, CTS3321, CTS3358, CTS3402, CTS3441, CTS3527, CTS3534, CTS3548, CTS3551, CTS358, CTS3607, CTS3622, CTS3649, CTS3650, CTS3654, CTS3697, CTS3775, CTS3848, CTS3943, CTS3984, CTS4259, CTS4465, CTS4862, CTS501, CTS5287, CTS5423, CTS5454, CTS5508, CTS5577, CTS5648, CTS570, CTS5936, CTS5979, CTS623, CTS6918, CTS6985, CTS7191, CTS7275, CTS7278, CTS7400, CTS7500, CTS7650, CTS7659, CTS7941, CTS8008, CTS8127, CTS8350, CTS8507, CTS8508, CTS8591, CTS8626, CTS8665, CTS8797, CTS8816, CTS8896, CTS9018, CTS903, CTS9108, CTS9510, CTS9515, CTS9539, CTS9596, CTS9733, CTS9739, CTS9754, CTS9779, F1050, F1209, F1224, F132, F1586, F184, F185, F2957, F3032, F313, F3159, F3166, F3185, F3194, F3197, F3337, F3561, F3564, F3644, F3650, F3692, F4099, F686, F719, F773, F82, F937, F989, IMS-JST002612, IMS-ST022448_3, IMS-ST022448_6, IMS-ST022449_3, IMS-ST022449_6, IMS-ST022451_2, K511, L1002, L1004, L1005, L1009, L1013, L1028, L104, L105, L1053, L1060, L1061, L1062, L1067, L1071, L108, L1089, L1090, L1095, L1098, L1099, L110, L1101, L1105, L1112, L1116, L1118, L112, L1120, L1121, L1123, L1124, L1125, L1127, L1129, L113, L1132, L1135, L1136, L1137, L114, L1142, L1143, L1145, L1150, L1155, L1179, L1185, L1120, L1225, L1235, L132, L1346, L1347, L1352, L1480, L1492, L15, L16, L168, L248, L268, L313, L350, L352, L413, L418, L438, L440, L449, L457, L468, L469, L498, L543, L58, L604, L721, L74, L741, L747, L76, L760, L768, L777, L779, L781, L819, L82, L821, L851, L875, L882, L962, L969, L970, L977, L986, L989, M168, M173, M198, M207, M213, M235, M251, M294, M299, M306, M417, M42, M420, M448, M449, M45, M459, M511, M512, M513, M514, M515, M516, M526, M74, M89, M9, M94, P108, P128, P131, P132, P133, P134, P135, P136, P138, P139, P14, P140, P141, P143, P145, P146, P148, P149, P151, P157, P158, P159, P160, P161, P163, P166, P187, P19_1, P207, P224, P225, P226, P228, P229, P230, P231, P232, P233, P234, P235, P236, P237, P238, P239, P242, P243, P244, P245, P27.1_2, P280, P281, P282, P283, P284, P285, P286, P294, P305, P316, PAGES00026, PAGES00081, PAGES00083, PF1345, PF1378, PF1389, PF1396, PF1402, PF1403, PF1405, PF1407, PF1408, PF1411, PF1412, PF1414, PF1415, PF1418, PF1419, PF2589, PF2590, PF2591, PF2608, PF2610, PF2611, PF2614, PF2615, PF2616, PF2619, PF2620, PF2621, PF2622, PF2626, PF2629, PF2631, PF2634, PF2635, PF2637, PF2640, PF2642, PF2647, PF2651, PF2653, PF2658, PF2660, PF2683, PF2685, PF288, PF2690, PF2694, PF2695, PF2700, PF2709, PF2716, PF2718, PF2722, PF2723, PF2729, PF2731, PF2732, PF2736, PF2737, PF2739, PF2742, PF2747, PF2748, PF2749, PF2752, PF2753, PF2754, PF2755, PF2756, PF2757, PF2758, PF2770, PF2772, PF2988, PF349, PF3494, PF3495, PF3496, PF3497, PF3500, PF4522, PF5459, PF5469, PF5477, PF5483, PF5488, PF5490, PF5498, PF5500, PF5503, PF5513, PF5861, PF5862, PF5863, PF5864, PF5869, PF5870, PF5871, PF5872, PF5876, PF5881, PF5882, PF5885, PF5886, PF5888, PF5889, PF5895, PF5908, PF5911, PF5914, PF5916, PF5919, PF5920, PF5936, PF5940, PF5943, PF5944, PF5945, PF5949, PF5951, PF5953, PF5954, PF5955, PF5956, PF5957, PF5958, PF5964, PF5965, PF5967, PF5970, PF5971, PF5975, PF5978, PF5980, PF5981, PF5982, PF5984, PF5985, PF5991, PF6013, PF6016, PF6047, PF6054, PF6056, PF6057, PF6066, PF6069, PF6071, PF6072, PF6073, PF6114, PF6116, PF6118, PF6121, PF6132, PF6133, PF6143, PF6153, PF6156, PF6158, PF6159, PF6160, PF6162, PF6163, PF6165, PF6168, PF6169, PF6170, PF6172, PF6179, PF6181, PF6194, PF6195, PF6201, PF6204, PF6211, PF6212, PF6213, PF6214, PF6215, PF6216, PF6217, PF6218, PF6220, PF6222, PF6230, PF6231, PF6233, PF6512, PF682, PF7514, PF7522, PF7527, PF7530, PF7531, PF7532, PF7534, PF7540, PF7542, PK1, PR1220, PR202, PR2119, PR5761, PR855, rs161492_1, V221, V238, V241, V250, Y2902, Y32, Y33, Y34, Y36, Y783.2, YSC0000057, YSC0000077, YSC0000081, YSC0000107, YSC0000108, YSC0000109, YSC0000110, YSC0000193, YSC0000350, YSC0000913, Z1331, Z280, Z282, Z5997, Z6030, Z6032. Фрагмент из списка снипов гаплогруппы A0-T (приведены 182 снипа из 587) Понятие «вилка» не стоит понимать буквально, А00 и А0-Т могли образоваться с разницей в десятки тысяч лет. По данным YFull, до начала А00 насчитывается 1684 снипа, до начала А0-Т – 587 снипов, но возраст обеих гаплогрупп почему-то выставлен как одинаковый, 235 тысяч лет назад. На такую повторяющуюся «дихотомию» в презентации данных YFull мы уже не раз обращали внимание. В любом случае, искать у нас, читателей Переформата, снипы гаплогруппы А00 бесполезно, нет их у нас, ни одного из тех 1684 снипов. Но важно то, что А0-Т образует очередную вилку, на гаплогруппы А0 и А1. Вторая вилка – расхождение А0-Т на А0 и А1. Потомки А0 сейчас живут в Африке, потомки А1 – мы с вами, читатели Переформата. У всех нас в цепочках снипов есть снипы субклада А1 (см. ниже), но это опять не значит, что наши предки вышли из Африки. Это опять промежуточные снипы, и никого с терминальными снипами А1 пока не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» никакого смысла тоже не имеет. По данным, приведенным YFull, к A0 ведет 899 снипов (это 129460 лет, если снип образуется в среднем раз в 144 года, как считали те же YFull), к А1 – 62 снипа (8900 лет), возраст А0 не выставлен, но для А1 выставлено 146300 лет «возраста». Опять загадка, как это может быть. Судя по этим данным, на протяжении 100 тысяч лет между А00 и А0-Т, гаплогрупп между ними вовсе не образовывалось, и к чему те 62 снипа относятся – непонятно. Но здесь важнее то, что А0 – это «африканская» гаплогруппа (опять по нынешнему месту жительства), а А1 – неафриканская. На схеме выше показано семь субкладов гаплогрупы А0, и снипов никаких из них у нас, читателей Переформата, нет, а снипы гаплогруппы А1 есть у нас всех (я не буду больше повторять, что это относится к обладателям Y-хромосомы, то есть к мужчинам, и что «читатель Переформата» относится к неафриканцам, потомкам гаплогруппы ВТ, которые не перебрались на жительство в Африку). Повторю, что «африканских» снипов А0 у нас нет, можно не искать, нет ни одного из 899 тех снипов. Не выходили наши предки из Африки, или, во всяком случае, не являются потомками ни А00, ни А0. Надо сказать, что в Африке носителей гаплогруппы А0 обнаружено немного. Scozzari et al (2012) указывали, что только после долгих поисков нашли трех (!) носителей гаплогруппы А0, все в Камеруне, и отмечали, что эта гаплогруппа имеет в Африке очень низкую частоту встречаемости. Ниже приведен список из 62 снипов гаплогруппы А1, из которых у меня есть L986, L989, L1002, L1009, L1112, V238, V241, V250. Остальных нет потому, что не все снипы у каждого определяются. Список снипов гаплогруппы A1 Понятие «вилка» не стоит понимать буквально, А00 и А0-Т могли образоваться с разницей в десятки тысяч лет. По данным YFull, до начала А00 насчитывается 1684 снипа, до начала А0-Т – 587 снипов, но возраст обеих гаплогрупп почему-то выставлен как одинаковый, 235 тысяч лет назад. На такую повторяющуюся «дихотомию» в презентации данных YFull мы уже не раз обращали внимание. В любом случае, искать у нас, читателей Переформата, снипы гаплогруппы А00 бесполезно, нет их у нас, ни одного из тех 1684 снипов. Но важно то, что А0-Т образует очередную вилку, на гаплогруппы А0 и А1. Вторая вилка – расхождение А0-Т на А0 и А1. Потомки А0 сейчас живут в Африке, потомки А1 – мы с вами, читатели Переформата. У всех нас в цепочках снипов есть снипы субклада А1 (см. ниже), но это опять не значит, что наши предки вышли из Африки. Это опять промежуточные снипы, и никого с терминальными снипами А1 пока не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» никакого смысла тоже не имеет. По данным, приведенным YFull, к A0 ведет 899 снипов (это 129460 лет, если снип образуется в среднем раз в 144 года, как считали те же YFull), к А1 – 62 снипа (8900 лет), возраст А0 не выставлен, но для А1 выставлено 146300 лет «возраста». Опять загадка, как это может быть. Судя по этим данным, на протяжении 100 тысяч лет между А00 и А0-Т, гаплогрупп между ними вовсе не образовывалось, и к чему те 62 снипа относятся – непонятно. Но здесь важнее то, что А0 – это «африканская» гаплогруппа (опять по нынешнему месту жительства), а А1 – неафриканская. На схеме выше показано семь субкладов гаплогрупы А0, и снипов никаких из них у нас, читателей Переформата, нет, а снипы гаплогруппы А1 есть у нас всех (я не буду больше повторять, что это относится к обладателям Y-хромосомы, то есть к мужчинам, и что «читатель Переформата» относится к неафриканцам, потомкам гаплогруппы ВТ, которые не перебрались на жительство в Африку). Повторю, что «африканских» снипов А0 у нас нет, можно не искать, нет ни одного из 899 тех снипов. Не выходили наши предки из Африки, или, во всяком случае, не являются потомками ни А00, ни А0. Надо сказать, что в Африке носителей гаплогруппы А0 обнаружено немного. Scozzari et al (2012) указывали, что только после долгих поисков нашли трех (!) носителей гаплогруппы А0, все в Камеруне, и отмечали, что эта гаплогруппа имеет в Африке очень низкую частоту встречаемости. Ниже приведен список из 62 снипов гаплогруппы А1, из которых у меня есть L986, L989, L1002, L1009, L1112, V238, V241, V250. Остальных нет потому, что не все снипы у каждого определяются. Список снипов гаплогруппы A1 Третья вилка – расхождение А1 на A1a и A1b. Потомки A1a сейчас живут в Африке и по всему миру (в частности, в ряде европейских стран и на Ближнем Востоке). Снипы А1b есть у всех нас, читатели Переформата. Но это опять не значит, что наши предки вышли из Африки. Это опять промежуточные снипы, и никого с терминальными снипами А1b пока не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» опять никакого смысла не имеет. По данным, приведенным YFull, к A1a ведет 495 снипов (это 71 тысяча лет, если снип образуется в среднем раз в 144 года), к А1b – 44 снипа (6300 лет), но почему-то им опять было выставлено по 132600 лет «возраста». Опять загадка, на каком основании так делается. Хотя носители гаплогруппы A1a разбросаны по всему миру (поэтому в любом случае это не «африканская» гаплогруппа, даже по такому условному критерию, как современное место жительства), их мало. И.Р. Рожанский уже приводил в своей статье «Африканцы на севере Европы. Африканцы ли?», что гаплогруппа A1a-M31 чрезвычайно редкая, и даже в Африке ее наличие исчисляется в лучшем случае 4.4% (в Западной Африке – Гвинея-Бисау, Гамбия, Сенегал, Мали, Нигер, Марокко), и 0.4% среди афроамериканцев. Этому же вторит статья Scozzari et al. (2012), что им удалось найти в Африке для геномных иследований только двоих (!) носителей A1a-M31. Ниже приведен список из 44 снипов гаплогруппы А1b, но в той системе, которую использует YFull. В классификации ISOGG в гаплогруппе A1b насчитывается 47 снипов, и ни один не пересекается по индексам с приведенными ниже. Из этих 47 снипов у меня есть L989, L1009, L1013, L1053, P108, V221. Остальных нет, потому что, как указывалось выше, не все снипы у каждого определяются. Список снипов гаплогруппы A1b (согласно классификации, используемой YFull) Третья вилка – расхождение А1 на A1a и A1b. Потомки A1a сейчас живут в Африке и по всему миру (в частности, в ряде европейских стран и на Ближнем Востоке). Снипы А1b есть у всех нас, читатели Переформата. Но это опять не значит, что наши предки вышли из Африки. Это опять промежуточные снипы, и никого с терминальными снипами А1b пока не выявлено, ни в Африке, ни вне Африки. Поэтому называть его «африканским» опять никакого смысла не имеет. По данным, приведенным YFull, к A1a ведет 495 снипов (это 71 тысяча лет, если снип образуется в среднем раз в 144 года), к А1b – 44 снипа (6300 лет), но почему-то им опять было выставлено по 132600 лет «возраста». Опять загадка, на каком основании так делается. Хотя носители гаплогруппы A1a разбросаны по всему миру (поэтому в любом случае это не «африканская» гаплогруппа, даже по такому условному критерию, как современное место жительства), их мало. И.Р. Рожанский уже приводил в своей статье «Африканцы на севере Европы. Африканцы ли?», что гаплогруппа A1a-M31 чрезвычайно редкая, и даже в Африке ее наличие исчисляется в лучшем случае 4.4% (в Западной Африке – Гвинея-Бисау, Гамбия, Сенегал, Мали, Нигер, Марокко), и 0.4% среди афроамериканцев. Этому же вторит статья Scozzari et al. (2012), что им удалось найти в Африке для геномных иследований только двоих (!) носителей A1a-M31. Ниже приведен список из 44 снипов гаплогруппы А1b, но в той системе, которую использует YFull. В классификации ISOGG в гаплогруппе A1b насчитывается 47 снипов, и ни один не пересекается по индексам с приведенными ниже. Из этих 47 снипов у меня есть L989, L1009, L1013, L1053, P108, V221. Остальных нет, потому что, как указывалось выше, не все снипы у каждого определяются. Список снипов гаплогруппы A1b (согласно классификации, используемой YFull) И вот здесь снипы гаплогруппы A1b приоткрывают загадку. Воспроизведем еще раз фрагмент лесенки снипов гаплогруппы A1: И вот здесь снипы гаплогруппы A1b приоткрывают загадку. Воспроизведем еще раз фрагмент лесенки снипов гаплогруппы A1:  Мы видим, что от нее вилкой отходят субклады A1a и A1b. Мы уже узнали, что А1а встречается в Европе, на Ближнем Востоке, в Африке, причем нет никаких данных, что он изначально был африканским. Субклад A1b встречается у всех неафриканцев как промежуточный, в цепочке снипов. Субкладов, нижестоящих от A1b, а это A1b1, A1b1a, A1b1a1, A1b1a1a, A1b1a1a1, A1b1a1a2, A1b1a1a2a, A1b1a1a2b – у неафриканцев нет, возможно, потому, что параллельно с A1b1 образовался субклад BT (по даным YFull примерно 126 тысяч лет назад), и A1b1 ушли в Африку, а ВТ остался вне Африки. То есть у неафриканцев имется основная линия A1b > BT, и потомки ее составляют все гаплогруппы от В до Т. И вдруг выясняется, что нижестоящая линия, в субкладе A1b1a1- M23, P3, обнаружилась в Южной Африке среди носителей койсанских языков. Но это – довольно далекий по цепочке вниз субклад, который, по-видимому, отошел от неафриканцев в древние времена. Иначе говоря, не неафриканцы отошли от африканцев, а наоборот, африканцы отошли от неафриканцев, мигрировав в Африку. Опять – не выходили наши предки из Африки. Было наоборот: современные африканцы – потомки неафриканцев. Четвертая вилка – расхождение A1b на A1b1 и BT. A1b1 и нисходящие субклады считаются африканскими – по нынешнему месту проживания его носителей, хотя родительская гаплогруппа A1b (как промежуточный субклад в цепочке снипов) насчитывает не менее 2-3 миллиардов неафриканцев, а нижеследующие субклады от A1b1 и ниже – обнаружены всего у нескольких африканцев. Вот и считайте, кто от кого произошел. Ответ ясен – африканцы от неафриканцев. Повторю – не выходили наши предки из Африки. Итак, потомки A1b1 сейчас живут в минимальном количестве в Африке, а потомки гаплогруппы ВТ – это все неафриканские гаплогруппы от В до Т. По данным YFull, к A1b1 ведут всего 26 снипов, к субкладу A1b1a ведут всего 2 снипа, к A1b1a1 – 11 снипов, ни одного из них у нас нет. Датировок ни к одному из нисходящих от A1b1 субкладов в списке YFull нет, и понятно, почему – их носители крайне редко встречаются, да и данных нет. Снипы ВТ есть у всех нас, читатели Переформата. Список снипов гаплогруппы A1b1 (согласно классификации, используемой YFull) Мы видим, что от нее вилкой отходят субклады A1a и A1b. Мы уже узнали, что А1а встречается в Европе, на Ближнем Востоке, в Африке, причем нет никаких данных, что он изначально был африканским. Субклад A1b встречается у всех неафриканцев как промежуточный, в цепочке снипов. Субкладов, нижестоящих от A1b, а это A1b1, A1b1a, A1b1a1, A1b1a1a, A1b1a1a1, A1b1a1a2, A1b1a1a2a, A1b1a1a2b – у неафриканцев нет, возможно, потому, что параллельно с A1b1 образовался субклад BT (по даным YFull примерно 126 тысяч лет назад), и A1b1 ушли в Африку, а ВТ остался вне Африки. То есть у неафриканцев имется основная линия A1b > BT, и потомки ее составляют все гаплогруппы от В до Т. И вдруг выясняется, что нижестоящая линия, в субкладе A1b1a1- M23, P3, обнаружилась в Южной Африке среди носителей койсанских языков. Но это – довольно далекий по цепочке вниз субклад, который, по-видимому, отошел от неафриканцев в древние времена. Иначе говоря, не неафриканцы отошли от африканцев, а наоборот, африканцы отошли от неафриканцев, мигрировав в Африку. Опять – не выходили наши предки из Африки. Было наоборот: современные африканцы – потомки неафриканцев. Четвертая вилка – расхождение A1b на A1b1 и BT. A1b1 и нисходящие субклады считаются африканскими – по нынешнему месту проживания его носителей, хотя родительская гаплогруппа A1b (как промежуточный субклад в цепочке снипов) насчитывает не менее 2-3 миллиардов неафриканцев, а нижеследующие субклады от A1b1 и ниже – обнаружены всего у нескольких африканцев. Вот и считайте, кто от кого произошел. Ответ ясен – африканцы от неафриканцев. Повторю – не выходили наши предки из Африки. Итак, потомки A1b1 сейчас живут в минимальном количестве в Африке, а потомки гаплогруппы ВТ – это все неафриканские гаплогруппы от В до Т. По данным YFull, к A1b1 ведут всего 26 снипов, к субкладу A1b1a ведут всего 2 снипа, к A1b1a1 – 11 снипов, ни одного из них у нас нет. Датировок ни к одному из нисходящих от A1b1 субкладов в списке YFull нет, и понятно, почему – их носители крайне редко встречаются, да и данных нет. Снипы ВТ есть у всех нас, читатели Переформата. Список снипов гаплогруппы A1b1 (согласно классификации, используемой YFull) К сводной гаплогруппе ВТ ведут 403 снипа, и многие из них есть у нас. У гаплогрупп категории А их, естественно, нет. Датировка ВТ по данным YFull – примерно 126 тысяч лет, хотя 403 снипа – это примерно 58 тысяч лет. Некоторые могут подумать, что эти снипы – от родительской гаплогруппы A1b, но та датирована той же компанией YFull примерно 133 тысячами лет назад, то есть всего на 7000 лет раньше. Опять остается неясным, к чему те 403 снипа относятся, и откуда тянутся. Самая загадка в том, как «параллельные» гаплогруппы A1b1 и BT, от одного общего предка A1b, и к которым идут 26 и 403 снипа, соответственно, могут оказаться с одинаковой датировкой – 126 тысяч лет. Ниже приведен частичный список снипов сводной гаплогруппы ВТ. Как уже отмечалось, многие из них есть у каждого неафриканского мужчины. У меня есть, например, L418, L604, L962, L970, L977, PF1408 и другие. Фрагмент из списка снипов гаплогруппы BT (приведены 166 снипов из 403) К сводной гаплогруппе ВТ ведут 403 снипа, и многие из них есть у нас. У гаплогрупп категории А их, естественно, нет. Датировка ВТ по данным YFull – примерно 126 тысяч лет, хотя 403 снипа – это примерно 58 тысяч лет. Некоторые могут подумать, что эти снипы – от родительской гаплогруппы A1b, но та датирована той же компанией YFull примерно 133 тысячами лет назад, то есть всего на 7000 лет раньше. Опять остается неясным, к чему те 403 снипа относятся, и откуда тянутся. Самая загадка в том, как «параллельные» гаплогруппы A1b1 и BT, от одного общего предка A1b, и к которым идут 26 и 403 снипа, соответственно, могут оказаться с одинаковой датировкой – 126 тысяч лет. Ниже приведен частичный список снипов сводной гаплогруппы ВТ. Как уже отмечалось, многие из них есть у каждого неафриканского мужчины. У меня есть, например, L418, L604, L962, L970, L977, PF1408 и другие. Фрагмент из списка снипов гаплогруппы BT (приведены 166 снипов из 403) Итак, первая часть этой статьи безоговорочно показывает, что «африканские ДНК-линии» совсем не древние, в противовес тому, чем нас пичкают популяционные генетики уже 30 лет. Если вести «древность» от общих предков с приматами, то африканские линии ничем не отличаются от неафриканских, имеют одну и ту же глубину. Если считать от времени жизни общих предков современных мужских популяций – по племенам или по территориям, то африканские ДНК-линии весьма мелкие, намного мельче, чем неафриканские. Если считать от образования гаплогрупп, то никто не знает, на каких территориях или континентах они образовались, поэтому присваивать им названия «африканские» – некорректно. Более того, носителей этих гаплогрупп, категории А, в Африке очень мало. К этому могут быть разные причины – прошли бутылочные горлышки популяции, вымерли, или прибыли в Африку относительно недавно – большого значения не имеет, они не «древние», в том смысле, который вкладывают попгенетики. В итоге имеем то, что изображено на заглавной иллюстрации поста. Так что с этим разделом закончили – нет в Африке «древних ДНК-линий». Но есть такое понятие, как совокупные ДНК-линии. Это тогда, когда сами ДНК-линии мелкие, но они – ошметки древних раскидистых деревьев гаплогрупп и субкладов. Мы видим только верхушки, но экстраполяция верхушек, в данном случае гаплотипов этих «ошметок», позволяет увидеть, когда жили общие предки этих разбросанных гаплотипов. Мы уже видели это выше – сравнение гаплотипов «мелких» популяций гаплогрупп А00 и А0 показывает, что они расходятся на астрономические расстояния. Сами «ошметки» имеют общих предков всего несколько сотен лет назад, но они расходятся на полмилиона лет, и отсюда следует, что их общие предки жили около 250 тысяч лет назад. В этом отношении африканские ДНК-линии отличаются от неафриканских. Рассмотрим это подробнее, в этом упрятана одна из основных загадок человечества. Загадка была впервые сформулирована в 2012 году, в статье (Klyosov, Rozhanskii, Advances in Anthropology, 2, No. 2, 80-86). Суть ее состояла в том, что все ДНК-линии современных гаплогрупп от В до Т, то есть неафриканских гаплогрупп, сходятся к общему предку, который жил 64000±6000 лет назад, хотя разрозненные линии африканских (по нынешнему месту жительства) гаплогрупп сходятся к общему предку 132 тысяч лет назад (хотя сами разрозненные линии датируются временами жизни общих предков всего лишь 5500, 5000 и 600 лет назад). При этом все неафриканские линии (включая гаплогруппу В) отстоят исключительно далеко от африканских линий, ясно показывая, что неафриканские линии не могли произойти от африканских. Например, базовые гаплотипы гаплогрупп А0 и В отстоят друг от друга на 270 тысяч лет (между ними 18 мутаций в «медленных» 22-маркерных гаплотипах; для сравнения, базовые гаплотипы R1a и R1b отстоят друг от друга всего на 8 мутаций, то есть примерно на 54 тысячи лет, и это помещает их общего предка на 27 тысяч лет назад), иллюстрация увеличивается по клику. Итак, первая часть этой статьи безоговорочно показывает, что «африканские ДНК-линии» совсем не древние, в противовес тому, чем нас пичкают популяционные генетики уже 30 лет. Если вести «древность» от общих предков с приматами, то африканские линии ничем не отличаются от неафриканских, имеют одну и ту же глубину. Если считать от времени жизни общих предков современных мужских популяций – по племенам или по территориям, то африканские ДНК-линии весьма мелкие, намного мельче, чем неафриканские. Если считать от образования гаплогрупп, то никто не знает, на каких территориях или континентах они образовались, поэтому присваивать им названия «африканские» – некорректно. Более того, носителей этих гаплогрупп, категории А, в Африке очень мало. К этому могут быть разные причины – прошли бутылочные горлышки популяции, вымерли, или прибыли в Африку относительно недавно – большого значения не имеет, они не «древние», в том смысле, который вкладывают попгенетики. В итоге имеем то, что изображено на заглавной иллюстрации поста. Так что с этим разделом закончили – нет в Африке «древних ДНК-линий». Но есть такое понятие, как совокупные ДНК-линии. Это тогда, когда сами ДНК-линии мелкие, но они – ошметки древних раскидистых деревьев гаплогрупп и субкладов. Мы видим только верхушки, но экстраполяция верхушек, в данном случае гаплотипов этих «ошметок», позволяет увидеть, когда жили общие предки этих разбросанных гаплотипов. Мы уже видели это выше – сравнение гаплотипов «мелких» популяций гаплогрупп А00 и А0 показывает, что они расходятся на астрономические расстояния. Сами «ошметки» имеют общих предков всего несколько сотен лет назад, но они расходятся на полмилиона лет, и отсюда следует, что их общие предки жили около 250 тысяч лет назад. В этом отношении африканские ДНК-линии отличаются от неафриканских. Рассмотрим это подробнее, в этом упрятана одна из основных загадок человечества. Загадка была впервые сформулирована в 2012 году, в статье (Klyosov, Rozhanskii, Advances in Anthropology, 2, No. 2, 80-86). Суть ее состояла в том, что все ДНК-линии современных гаплогрупп от В до Т, то есть неафриканских гаплогрупп, сходятся к общему предку, который жил 64000±6000 лет назад, хотя разрозненные линии африканских (по нынешнему месту жительства) гаплогрупп сходятся к общему предку 132 тысяч лет назад (хотя сами разрозненные линии датируются временами жизни общих предков всего лишь 5500, 5000 и 600 лет назад). При этом все неафриканские линии (включая гаплогруппу В) отстоят исключительно далеко от африканских линий, ясно показывая, что неафриканские линии не могли произойти от африканских. Например, базовые гаплотипы гаплогрупп А0 и В отстоят друг от друга на 270 тысяч лет (между ними 18 мутаций в «медленных» 22-маркерных гаплотипах; для сравнения, базовые гаплотипы R1a и R1b отстоят друг от друга всего на 8 мутаций, то есть примерно на 54 тысячи лет, и это помещает их общего предка на 27 тысяч лет назад), иллюстрация увеличивается по клику.  Ясно, что если между гаплогруппами А0 и В 270 тысяч лет, то вторая никак не могла образоваться от первой в течение всего периода существования Homo sapiens, не говоря о 70 тысячах лет назад, которые попгенетики отводят для времени образования гаплогруппы В из А. Это аналогично примеру выше для R1a и R1b, в котором 54 тысячи лет расстояния между ними никак не могут допустить, что одна образовалась из другой; они обе образовались от одного общего предка, которым в данном случае являлась гаплогруппа R1. Гаплогруппа А0, далеко отстоящая от гаплогруппы В, здесь не исключение. Подобные огромные расстояния отделяют гаплогруппу В от любых гаплотипов в субкладах категории А, например, 250 тысяч лет от A0a-L981, 280 тысяч лет от A1b-M32, 310 тысяч лет от европейского кластера гаплогруппы A1a-M31, 360 тысяч лет от другого кластера той же гаплогруппы, 320 тысяч лет от арабского кластера той же гаплогруппы, 350 тысяч лет от A0a-V150, 356 тысяч лет от A0a-L979, 346 тысяч лет от A0a1a-V151, и так далее. Самое большое расстояние отделяет гаплогруппу В от А00 – 450 тысяч лет, то есть их общий предок жил примерно 225 тысяч лет назад, на утренней заре Homo sapiens. Такая же картина наблюдается и для гаплотипов всех гаплогрупп от В до Т. Например, растояние от гаплогруппы С до А0-L896 составляет 357 тысяч лет, до A0a1-V150 – 300 тысяч лет, и так далее. Но расстояние от В до С составляет всего 120 тысяч лет, то есть их общий предок жил примерно 60 тысяч лет назад. Это находится в полном соответствии с картиной, что гаплогруппа В образует единый кластер с гаплогруппами от В до Т, и не является по происхождению «африканской» гаплогруппой. Видимо, уже всем ясно, что гаплогруппа В не образовалась из какой-либо гаплогруппы категории А (поскольку нет гаплогруппы А как таковой, есть А00, А0-Т, А0, А1, А1a, A1b и так далее), а все они ведут свое начало от общего предка. Этот общий предок жил примерно 160 тысяч лет назад, как показано в упомянутой выше статье (Klyosov, Rozhanskii, Advances in Anthropology, 2, No. 2, 80-86). Если исходить из датировок, приведенных на сайте YFull, то основатели гаплогрупп А00 и A0-T жили раньше этого времени, причем гаплогруппа А00 осталась в известном смысле тупиковой, нисходящие от нее субклады и прочие ДНК-линии неизвестны. A0-T прошла своими снипами как к африканцам, так и к неафриканцам, как показано выше, поскольку образовалась еще до разделения на африканскую и неафриканскую ветви. Разделение на эти ветви соответствует временам непосредственно до образования гаплогрупп А0 и А1, причем А0 ушла в Африку, А1 осталась вне Африки. Впоследствии носители многих гаплогрупп мигрировали в Африку, например, A1a и нижестоящие субклады, A1b1 и нижестоящие субклады, поэтому их снипов у нас нет, как описано выше. Все это описано в целом в упомянутой выше статье (Klyosov, Rozhanskii, Advances in Anthropology, 2012), которая, кстати, набрала рекордное количество просмотров (около 90 тысяч) и скачиваний (около 20 тысяч), но академическое сообщество предпочло ее не заметить. Слишком многое она меняет в представлениях. Основная иллюстрация в той статье следующая: Ясно, что если между гаплогруппами А0 и В 270 тысяч лет, то вторая никак не могла образоваться от первой в течение всего периода существования Homo sapiens, не говоря о 70 тысячах лет назад, которые попгенетики отводят для времени образования гаплогруппы В из А. Это аналогично примеру выше для R1a и R1b, в котором 54 тысячи лет расстояния между ними никак не могут допустить, что одна образовалась из другой; они обе образовались от одного общего предка, которым в данном случае являлась гаплогруппа R1. Гаплогруппа А0, далеко отстоящая от гаплогруппы В, здесь не исключение. Подобные огромные расстояния отделяют гаплогруппу В от любых гаплотипов в субкладах категории А, например, 250 тысяч лет от A0a-L981, 280 тысяч лет от A1b-M32, 310 тысяч лет от европейского кластера гаплогруппы A1a-M31, 360 тысяч лет от другого кластера той же гаплогруппы, 320 тысяч лет от арабского кластера той же гаплогруппы, 350 тысяч лет от A0a-V150, 356 тысяч лет от A0a-L979, 346 тысяч лет от A0a1a-V151, и так далее. Самое большое расстояние отделяет гаплогруппу В от А00 – 450 тысяч лет, то есть их общий предок жил примерно 225 тысяч лет назад, на утренней заре Homo sapiens. Такая же картина наблюдается и для гаплотипов всех гаплогрупп от В до Т. Например, растояние от гаплогруппы С до А0-L896 составляет 357 тысяч лет, до A0a1-V150 – 300 тысяч лет, и так далее. Но расстояние от В до С составляет всего 120 тысяч лет, то есть их общий предок жил примерно 60 тысяч лет назад. Это находится в полном соответствии с картиной, что гаплогруппа В образует единый кластер с гаплогруппами от В до Т, и не является по происхождению «африканской» гаплогруппой. Видимо, уже всем ясно, что гаплогруппа В не образовалась из какой-либо гаплогруппы категории А (поскольку нет гаплогруппы А как таковой, есть А00, А0-Т, А0, А1, А1a, A1b и так далее), а все они ведут свое начало от общего предка. Этот общий предок жил примерно 160 тысяч лет назад, как показано в упомянутой выше статье (Klyosov, Rozhanskii, Advances in Anthropology, 2, No. 2, 80-86). Если исходить из датировок, приведенных на сайте YFull, то основатели гаплогрупп А00 и A0-T жили раньше этого времени, причем гаплогруппа А00 осталась в известном смысле тупиковой, нисходящие от нее субклады и прочие ДНК-линии неизвестны. A0-T прошла своими снипами как к африканцам, так и к неафриканцам, как показано выше, поскольку образовалась еще до разделения на африканскую и неафриканскую ветви. Разделение на эти ветви соответствует временам непосредственно до образования гаплогрупп А0 и А1, причем А0 ушла в Африку, А1 осталась вне Африки. Впоследствии носители многих гаплогрупп мигрировали в Африку, например, A1a и нижестоящие субклады, A1b1 и нижестоящие субклады, поэтому их снипов у нас нет, как описано выше. Все это описано в целом в упомянутой выше статье (Klyosov, Rozhanskii, Advances in Anthropology, 2012), которая, кстати, набрала рекордное количество просмотров (около 90 тысяч) и скачиваний (около 20 тысяч), но академическое сообщество предпочло ее не заметить. Слишком многое она меняет в представлениях. Основная иллюстрация в той статье следующая:  Номенклатура в этой иллюстрации несколько устаревшая, в ней нет гаплогруппы А00, которая в 2012 году была неизвестной, но суть ее остается правильной. Гаплогруппа альфа – это условная гаплогруппа, от которой пошло разделение эволюционного Y-хромосомного дерева человечества на африканскую (слева) и неафриканскую линии. В настоящее время все гаплогруппы от В до Т, то есть гаплогруппы неафриканского происхождения, сходятся к условной же бета-гаплогруппе, которой на самом деле является сводная гаплогруппа ВТ. Загадка – куда делась связка между альфа- и бета-гаплогруппой, протяженностью в сто тысяч лет? И, кстати, куда делись африканские ДНК-линии, которые на левой части диаграммы полностью экстраполированы, от общих предков всего от тысячи до нескольких тысяч лет давности, вглубь времен? Со времени публикации той статьи прошло уже более трех лет, в базах данных и в Проектах с тех пор накопилось много гаплотипов, в том числе протяженных, 111-маркерных, и появилась возможность проверить количественные выводы статьи. То, что расхождение африканской и неафриканской ДНК-линий произошло примерно 160 тысяч лет назад, мы уже проверили. Перейдем ко времени жизни общего предка всех неафриканских гаплогрупп – от В до Т. Ниже – дерево гаплотипов этих гаплогрупп. Оно построено по 111-маркерным гаплотипам, и сами гаплогруппы показаны по окружности дерева. Для каждой из 18 гаплогрупп использовали 10-25 гаплотипов, взятых произвольно из выборки, всего 403 гаплотипа nd 111-маркерном формате. Гаплогруппы категории А на дереве не оставили, так как они явно «вылезали» из кругового дерева, поскольку, как мы уже показали, они удалены по мутациям от всех других гаплогрупп. Номенклатура в этой иллюстрации несколько устаревшая, в ней нет гаплогруппы А00, которая в 2012 году была неизвестной, но суть ее остается правильной. Гаплогруппа альфа – это условная гаплогруппа, от которой пошло разделение эволюционного Y-хромосомного дерева человечества на африканскую (слева) и неафриканскую линии. В настоящее время все гаплогруппы от В до Т, то есть гаплогруппы неафриканского происхождения, сходятся к условной же бета-гаплогруппе, которой на самом деле является сводная гаплогруппа ВТ. Загадка – куда делась связка между альфа- и бета-гаплогруппой, протяженностью в сто тысяч лет? И, кстати, куда делись африканские ДНК-линии, которые на левой части диаграммы полностью экстраполированы, от общих предков всего от тысячи до нескольких тысяч лет давности, вглубь времен? Со времени публикации той статьи прошло уже более трех лет, в базах данных и в Проектах с тех пор накопилось много гаплотипов, в том числе протяженных, 111-маркерных, и появилась возможность проверить количественные выводы статьи. То, что расхождение африканской и неафриканской ДНК-линий произошло примерно 160 тысяч лет назад, мы уже проверили. Перейдем ко времени жизни общего предка всех неафриканских гаплогрупп – от В до Т. Ниже – дерево гаплотипов этих гаплогрупп. Оно построено по 111-маркерным гаплотипам, и сами гаплогруппы показаны по окружности дерева. Для каждой из 18 гаплогрупп использовали 10-25 гаплотипов, взятых произвольно из выборки, всего 403 гаплотипа nd 111-маркерном формате. Гаплогруппы категории А на дереве не оставили, так как они явно «вылезали» из кругового дерева, поскольку, как мы уже показали, они удалены по мутациям от всех других гаплогрупп.  Видно, что все гаплогруппы от В до Т образуют достаточно единую систему ДНК-линий (выраженную здесь через 111-маркерные гаплотипы), так что про некую «африканскую гаплогруппу В» говорить не приходится. Она такая же «автохтонная африканская», как R1b – «автохтонная европейская». Это застарелая болезнь попгенетиков – то, что видят сейчас, значит, так было всегда. Болезнь называется «отсутствие научного подхода», или «отсутствие научной школы», что, впрочем, одно и то же, а если по-русски, то «думать не умеют». 403 гаплотипа в 111-маркерном формате, то есть почти 45 тысяч аллелей – это крепкий орешек, если считать вручную. Более того, за десятки тысяч лет в них накапливаются десятки тысяч мутаций, помимо того, огромное количество возвратных мутаций, что делает 111-маркерную систему такой древности неустойчивой, и неприемлемой для расчетов, поскольку датировки значительно занижаются. Поэтому для данных случаев разработана 22-маркерная панель из самых «медленных» маркеров. Они настолько устойчивы во времени, что для всех 403 гаплотипов за десятки тысяч лет маркер DYS472 вообще не мутировал ни разу, DYS436 – всего 30 раз, DYS590 – 42 раза, и так далее. Сравните – маркер DYS390 за это же время должен мутировать 2270 раз, то есть в среднем 5.6 раз на гаплотип. При таких частотах мутации расчет вклада обратных мутаций уже практически невозможен, система нестабильна. А вот «медленная» 22-маркерная панель с малыми скоростями мутаций позволяет проводить расчеты на десятки и сотни тысяч лет вглубь, и даже на миллионы лет, как было показано выше на примере расчета времени жизни общих предков человека и шимпанзе. Более того, разработанный калькулятор Килина-Клёсова проводит расчеты по каждому маркеру отдельно, используя квадратичный метод, и поэтому не требует введения поправок на возвратные мутации. В итоге ДНК-генеалогия получила незаменимый метод расчета массивных серий гаплотипов в любом формате и любой древности общего предка (Килин и Клёсов, Вестник Академии ДНК-генеалогии, 2015, т. 8, № 3, 321-375, http://dna-academy.ru/kilin-klyosov/). Расчет дерева из 403 гаплотипов, приведенного выше, показал, что общий предок гаплогрупп от В до Т жил примерно 63 тысячи лет назад (результат без округления – 63036 лет, что, конечно, с такой точностью совершенно излишне). Это почти точно совпадает с рассчитанной ранее (Klyosov and Rozhanskii, 2012) величиной 64±6 тысяч лет. Теперь осталось подойти к решению сформулированной в той статье загадки – повторим ее опять: куда же делись предки Y-хромосомных линий в Африке ранее 5-10 тысяч лет назад, и у неафриканцев ранее 64±6 тысяч лет назад? Причем у африканских линий группы А можно косвенным путем, а именно при сравнении протяженных гаплотипов с «неглубокими» общими предками, достичь времен жизни общих предков 132 тысячи лет назад, а у неафриканцев, как их гаплотипы ни комбинировать и ни сравнивать, глубже 64±6 тысяч лет уйти не получается. Более древних общих предков как корова языком слизнула. Что же это за корова была такая? Некоторые предположения в цитированной выше статье сделаны были – упомянуто возможное последствие извержения вулкана Тоба в Индонезии, около 70 тысяч лет назад. Не исключено было резкое похолодание, возможно, как результат извержения того же вулкана, причем, по мнению специалистов, ранг катастрофичности глобального похолодания намного выше, чем извержения вулкана, но это все оставалось довольно абстрактными предположениями. И вообще – чтобы извержение вулкана в Индонезии привело к гибели почти всех людей на планете – это кажется маловероятным, но все это «по понятиям». Нужно было мнение специалиста, пусть мнение и спорное, неоднозначное, но главное – подтвержденное конкретными данными. И такой специалист по данной проблеме есть. Знакомьтесь – Валерий Павлович Юрковец, профессиональный геофизик, палеоклиматолог, специалист в области влияния климатических катастроф, в том числе импактного характера, на миграции древнего человека, член Академии ДНК-генеалогии с начала ее образования несколько лет назад. Продолжение следует… Анатолий А. Клёсов,доктор химических наук, профессор Перейти к авторской колонке Видно, что все гаплогруппы от В до Т образуют достаточно единую систему ДНК-линий (выраженную здесь через 111-маркерные гаплотипы), так что про некую «африканскую гаплогруппу В» говорить не приходится. Она такая же «автохтонная африканская», как R1b – «автохтонная европейская». Это застарелая болезнь попгенетиков – то, что видят сейчас, значит, так было всегда. Болезнь называется «отсутствие научного подхода», или «отсутствие научной школы», что, впрочем, одно и то же, а если по-русски, то «думать не умеют». 403 гаплотипа в 111-маркерном формате, то есть почти 45 тысяч аллелей – это крепкий орешек, если считать вручную. Более того, за десятки тысяч лет в них накапливаются десятки тысяч мутаций, помимо того, огромное количество возвратных мутаций, что делает 111-маркерную систему такой древности неустойчивой, и неприемлемой для расчетов, поскольку датировки значительно занижаются. Поэтому для данных случаев разработана 22-маркерная панель из самых «медленных» маркеров. Они настолько устойчивы во времени, что для всех 403 гаплотипов за десятки тысяч лет маркер DYS472 вообще не мутировал ни разу, DYS436 – всего 30 раз, DYS590 – 42 раза, и так далее. Сравните – маркер DYS390 за это же время должен мутировать 2270 раз, то есть в среднем 5.6 раз на гаплотип. При таких частотах мутации расчет вклада обратных мутаций уже практически невозможен, система нестабильна. А вот «медленная» 22-маркерная панель с малыми скоростями мутаций позволяет проводить расчеты на десятки и сотни тысяч лет вглубь, и даже на миллионы лет, как было показано выше на примере расчета времени жизни общих предков человека и шимпанзе. Более того, разработанный калькулятор Килина-Клёсова проводит расчеты по каждому маркеру отдельно, используя квадратичный метод, и поэтому не требует введения поправок на возвратные мутации. В итоге ДНК-генеалогия получила незаменимый метод расчета массивных серий гаплотипов в любом формате и любой древности общего предка (Килин и Клёсов, Вестник Академии ДНК-генеалогии, 2015, т. 8, № 3, 321-375, http://dna-academy.ru/kilin-klyosov/). Расчет дерева из 403 гаплотипов, приведенного выше, показал, что общий предок гаплогрупп от В до Т жил примерно 63 тысячи лет назад (результат без округления – 63036 лет, что, конечно, с такой точностью совершенно излишне). Это почти точно совпадает с рассчитанной ранее (Klyosov and Rozhanskii, 2012) величиной 64±6 тысяч лет. Теперь осталось подойти к решению сформулированной в той статье загадки – повторим ее опять: куда же делись предки Y-хромосомных линий в Африке ранее 5-10 тысяч лет назад, и у неафриканцев ранее 64±6 тысяч лет назад? Причем у африканских линий группы А можно косвенным путем, а именно при сравнении протяженных гаплотипов с «неглубокими» общими предками, достичь времен жизни общих предков 132 тысячи лет назад, а у неафриканцев, как их гаплотипы ни комбинировать и ни сравнивать, глубже 64±6 тысяч лет уйти не получается. Более древних общих предков как корова языком слизнула. Что же это за корова была такая? Некоторые предположения в цитированной выше статье сделаны были – упомянуто возможное последствие извержения вулкана Тоба в Индонезии, около 70 тысяч лет назад. Не исключено было резкое похолодание, возможно, как результат извержения того же вулкана, причем, по мнению специалистов, ранг катастрофичности глобального похолодания намного выше, чем извержения вулкана, но это все оставалось довольно абстрактными предположениями. И вообще – чтобы извержение вулкана в Индонезии привело к гибели почти всех людей на планете – это кажется маловероятным, но все это «по понятиям». Нужно было мнение специалиста, пусть мнение и спорное, неоднозначное, но главное – подтвержденное конкретными данными. И такой специалист по данной проблеме есть. Знакомьтесь – Валерий Павлович Юрковец, профессиональный геофизик, палеоклиматолог, специалист в области влияния климатических катастроф, в том числе импактного характера, на миграции древнего человека, член Академии ДНК-генеалогии с начала ее образования несколько лет назад. Продолжение следует… Анатолий А. Клёсов,доктор химических наук, профессор Перейти к авторской колонке Читайте другие статьи на Переформате: pereformat.ru Секвенирование древней ДНК Википедия ДНК, выделенная из древнеегипетской мумии возрастом 4000 лет ДНК, выделенная из древнеегипетской мумии возрастом 4000 лет Секвенирование древней ДНК — определение нуклеотидной последовательности (секвенирование, от лат. sequentum — последовательность) применительно к молекулам ДНК, извлечённым из древних биологических образцов, таких как палеонтологические и археологические находки, мумифицированные останки, засохшие остатки растений, копролиты. Анализ нуклеотидных последовательностей, полученных секвенированем древней ДНК, позволяют установить филогенетические отношения между видами и проверять гипотезы о связи изменений в окружающей среде и эволюционных изменений популяций, а также предоставляют информацию для калибровки молекулярных часов[1]. При работе с древней ДНК исследователи сталкиваются со множеством проблем, связанных с сохранностью образцов. ДНК может деградировать с течением времени, химически модифицироваться. Микроорганизмы, участвующие в разложении останков, не только нарушают целостность тканей, но и вносят в образец собственную ДНК, тем самым усложняя процесс выделения древней ДНК и биоинформатического анализа полученных данных. Такие методы, как секвенирование нового поколения и обогащение ДНК-библиотек путём гибридизации позволяют заметно увеличить количество получаемых из образцов информации. Проведен анализ ДНК ряда древних животных, в том числе мамонта и пещерного медведя. Анализ ДНК из человеческих останков позволил выделить новую группу древних людей — денисовцев, а также выявить детали происхождения современных этнических групп. Ряд открытий был сделан в результате анализа древней ДНК болезнетворных микроорганизмов: произведен анализ генома чумной палочки из лондонских захоронений XIV века и гриба фитофторы из образцов XIX века. История Исследования древней ДНК начались в 1984 году с секвенирования фрагмента митохондриальной ДНК (мтДНК) вымершей во второй половине XIX квагги[2], подвида бурчелловой зебры. Было обнаружено, что ДНК не только сохраняется на протяжении более полутора веков, но также может быть частично выделена и секвенирована. Вскоре после этого Сванте Паабо секвенировал образцы, полученные из человеческих мумий.[3][4] В этих исследованиях ученый использовал бактериальное клонирование для амплификации фрагментов ДНК. Оказалось, что большая часть ДНК в образцах имеет бактериальное или грибное происхождение, а поддающаяся амплификации эндогенная ДНК состоит в основном из коротких повреждённых фрагментов многокопийных локусов (например, мтДНК) и составляет малую часть исследованной ДНК. Изобретение полимеразной цепной реакции (ПЦР) позволило амплифицировать даже немногочисленные сохранившиеся фрагменты ДНК и дало толчок к развитию этой области, однако также увеличило чувствительность результатов к контаминации[5]. Области применения Исследование древней ДНК имеет важное значение для таких областей науки как генетика, палеозоология, палеоэпидемология, антропология (особенно палеоантропология) и археология. Также эта область стала частью методологии палеогенетики. Анализ древней ДНК может использоваться для оценки эволюционной близости таксономических групп давно вымерших или сильно изменившихся организмов, конретные родственные связи которых выяснить иными способами крайне затруднительно. Особое внимание здесь уделяется областям высокой изменчивости — участкам ДНК, где мутации происходят часто. В частности, это короткие тандемные повторы (STR) и однонуклеотидные полиморфизмы (SNP). Анализ митохондриальной ДНК дает более точную информацию в контексте этого метода, так как митохондриальная ДНК имеет гораздо большее количество копий по сравнению с ядерной ДНК (около 1000 копий митохондриальных ДНК и 2 копии ядерной ДНК на клетку)[6]. Анализ древней ДНК также позволяет устанавливать половую принадлежность скелетных останков представителей видов, женские и мужские особи которых отличаются набором половых хромосом, что особенно важно, когда другие методы, например антропометрия, не могут дать точного ответа[7]. Данный метод находит свое применение и в контексте палеоэпидемологии. Диагностику заболеваний по скелетным останкам и изучение древних пандемий крайне сложно проводить без анализа ДНК возбудителей, сохранившейся в останках больных. Такие исследования стали возможны только в 1990 годах, что позволило прослеживать как эволюцию инфекционных бактериальных штаммов и вирусов, так и их распространение, а также сопоставлять детектируемые по останкам симптомы болезни с современными знаниями о конкретном заболевании[8][9]. Технические сложности Деградация ДНК Обычно после смерти организма ДНК расщепляется эндогенными нуклеазами. Этого не происходит, если нуклеазы оказываются быстро разрушены или инактивированы, например, вследствие обезвоживания останков, низких температур или большой концентрации соли.[10] Даже в этом случае ДНК со временем повреждается в результате случайного гидролиза или окисления. К гидролитическим повреждениям относятся разрушение фосфатного остова цепи, депуринизация (англ.) (соответствующая позиция остается без азотистого основания) и дезаминирование. Чаще происходит дезаминирование цитозина в урацил, метилированный цитозин (5-метил-цитозин) дезаминируется в тимин; реже аденин превращается в гипоксантин, который комплементарен цитозину, а не тимину, что ведет к неправильному прочтению при секвенировании. Эти повреждения происходят и в живых клетках, однако там они устраняются в процессе репарации. Кроме того, возникают поперечные сшивки между цепями спирали ДНК из-за алкилирования или сшивки ДНК с различными молекулами в результате реакции Майяра. Как и разрывы цепи, поперечные сшивки мешают амплификации при ПЦР[10][5]. Перед секвенированием древнюю ДНК подвергают специальной обработке, чтобы удалить продукты дезаминирования и поперечные сшивки. Средняя длина амплифицируемых фрагментов древней ДНК часто не превосходит 100 пар нуклеотидов, причём для находок из одного места раскопок средняя длина фрагментов убывает с увеличением возраста находки. Современные протоколы секвенирования древней ДНК учитывают эту особенность; в частности, вместо отжига праймеров на фрагменты ДНК к концам фрагментов присоединяются адаптеры, а праймерами служат олигонуклеотиды, комплементарные адаптерам[11]. При низких температурах деградация ДНК идет медленнее, что обеспечивает хорошую сохранность ДНК в образцах, найденных в районе вечной мерзлоты. Однако считается, что даже в идеальных условиях ДНК не может сохраняться на протяжении более 1 млн лет[10][5]. Примесь чужеродной ДНК Часто вследствие загрязнения, или контаминации, лишь малая доля ДНК в образце имеет эндогенное происхождение. Лучшие в этом отношении образцы содержат до 90 % эндогенной ДНК.[1] Археологические образцы всегда содержат некоторое количество ДНК бактерий и грибов, которые колонизируют останки, пока те лежат в земле. Кроме того, в процессе исследования древней ДНК в образец может попасть человеческая или микробная ДНК, присутствующая в любой лаборатории. В отличие от древней ДНК современная хорошо амплифицируется при ПЦР. Продукты ПЦР, содержащие амплифицированную современную ДНК, могут распространиться по лаборатории и увеличить таким образом степень контаминации[1][5]. Для профилактики лабораторной контаминации рекомендуется продолжительная обработка оборудования ультрафиолетом или кислотой и соблюдение требований протокола работы в ПЦР-лабораториях. Исследование древней ДНК не должно проводиться в лаборатории, где ранее изучались современные образцы, особенно близких видов[5]. Многие археологические образцы, особенно найденные до появления современных молекулярно-биологических методов, подверглись контаминации в процессе извлечения и исследования. Если образец был найден несколько десятилетий назад, человеческая ДНК, которой он загрязнен, может обладать всеми признаками древней ДНК. Кости и зубы имеют пористую поверхность, что делает проблематичной их очистку от современной ДНК. Волосы оказываются в этом отношении предпочтительнее: содержащаяся в них ДНК малодоступна для бактерий, а гидрофобная поверхность позволяет очистить их перед извлечением ДНК[1]. В этом случае совпадение последовательностей, полученных в результате независимого исследования разных частей образца (например, бедра и зубов) в разных лабораториях свидетельствует в пользу истинности результата[5]. В случае человеческих останков контаминация современной человеческой ДНК может приводить к ошибкам при исследовании филогении и популяционной генетики. Существует статистический метод, позволяющий отличать древнюю ДНК от современной по паттерну дезаминирования[12][13]. Если известно, что образец ДНК принадлежит женщине, можно картировать отсеквенированные последовательности на Y-хромосому и детектировать таким образом попадание в образец мужской ДНК[14]. Если исследуется ДНК нечеловеческого происхождения, очевидным способом проверки на контаминацию является картирование полученных при секвенировании чтений на человеческий геном, а также на геномы других организмов в случае, если они могли быть источниками контаминации. При секвенировании древней бактериальной ДНК проблема состоит также в том, что известны геномы далеко не всех существующих ныне бактерий, так что из того, что полученная последовательность не входит в известные бактериальные геномы, нельзя заключить, что она принадлежит древней бактерии, а не получена в результате контаминации[5]. Вставки мтДНК и хпДНК в хромосомах Вставки митохондриальной и пластидной ДНК часто встречаются в ядерной ДНК. При исследовании древних образцов ДНК нельзя выделить ДНК органелл, поэтому эти вставки могут быть источником ошибок, так как их не всегда легко отличить по последовательности от настоящей мтДНК или хпДНК. Если такие вставки будут приняты за ДНК органелл, это может исказить результаты исследования филогении или популяционной генетики[5]. Методы обработки древней ДНК В качестве источника древней ДНК часто стараются использовать сохранившиеся части тела, не представляющие большого интереса с точки зрения анатомического строения. Из-за описанных выше процессов в большинстве древних образцов содержание интересующей исследователей древней ДНК очень мало — всего около 1 %. Это количество сильно варьирует в зависимости от природы образца. Последние исследования показывают, что значительно больший выход эндогенной ДНК из останков предков человека достигается при выделении материала из зубов и каменистой части височной кости.[15] Первым шагом является измельчение образца и выделение ДНК. В случае костных остатков используют пескоструйные пистолеты или специализированные дрели для первичной фрагментации. Далее частицы ещё более измельчаются (до порошкообразного состояния) на перемешивающих мельницах. Порошок последовательно обрабатывается набором реагентов и подвергается центрифугированию для очистки ДНК от минералов и прочих примесей[16]. Следующий этап — изготовление ДНК-библиотек и их обогащение путем гибридизации[17]. Непосредственное получение нуклеотидной последовательности осуществляется путем секвенирования нового поколения. В 2012 году в статье журнала Methods of Molecular Biology были представлены возможные методы выделения древней ДНК из сохранившихся растительных образцов. Как и в случае с древней ДНК животных, палеорастительные образцы доходят в сохранном виде до нашего времени зачастую из-за охлаждения во время масштабных оледенений[18]. «Допотопная» ДНК «Допотопной» (англ. antediluvian) была названа ДНК старше 1 миллиона лет. Название было предложено в 1993 году Томасом Линдалом (Tomas Lindahl) в обзоре в Nature[19]. В 1990-е годы появились сообщения о секвенировании ДНК, сохранившейся на протяжении миллионов лет в ископаемых остатках растений, костях динозавров и инклюзах в янтаре. Некоторые из этих результатов с большой вероятностью являются следствием контаминации, другие оказались невоспроизводимы.[10][5] Например, в ответ на публикацию в Science о секвенировании фрагмента митохондриальной ДНК из кости динозавра мелового периода (80 млн лет назад), вышли заметки, в одной из которых было указано, что при построении филогенетического дерева последовательность из кости динозавра кластеризуется с человеческой, а не с ортологичным участком из мтДНК птиц или крокодила, что свидетельствует о большой вероятности контаминации[20]; в другой заметке высказывалось предположение[21], что последовательность, приписанная динозавру, — это на самом деле древняя вставка мтДНК в человеческой хромосоме, попавшей в образец[22]. ДНК животных плейстоцена Хорошо сохранившиеся образцы позволяют частично, а в некоторых случаях даже полностью секвенировать ядерный геном. В 2008 году была секвенирована ДНК, полученная из шерсти двух мамонтов, умерших около 20 и 59 тысяч лет назад. Картирование на черновую сборку генома африканского слона позволило оценить долю эндогенной ДНК в этих образцах в 90 % и 58 % соответственно; большую часть загрязнения в обоих случаях составили бактериальная ДНК и последовательности, происхождение которых определить не удалось. Полученные данные позволили оценить время расхождения между мамонтом и африканским слоном в 7,5 млн лет, а время расхождения между двумя линиями мамонта, для которых были взяты образцы, — в 1,5-2 млн лет. При этом секвенированная ДНК мамонтов совпадает со слоновьей на 99,41 % на нуклеотидном уровне и 99,78 % на аминокислотном уровне (то есть различие составляет примерно 1 остаток на белок). Принадлежность М4 и М25 к разным кладам была установлена ранее на основе секвенирования митохондриальной ДНК, и новая оценка на время расхождения согласуется с оценкой по мтДНК и уточняет последнюю. Одной из целей секвенирования мамонта было определение функционально важных аминокислотных различий между мамонтом и слоном. Были отобраны 92 различия между этими видами, которые могут иметь функциональное значение, и часть из которых, возможно, находилась под положительным отбором[23]. В одном из более ранних исследований в качестве образцов были взяты хорошо сохранившиеся при низких температурах останки восьми мамонтов из Музея мамонта в Хатанге. Для лучшего из отобранных образцов 45,4 % фрагментов ДНК выравнивались на сборку генома слона, при этом сходство между ДНК слона и мамонта составило 98,55 % без поправки на увеличение числа различий из-за дезаминации[24]. В 2013 году была получена черновая версия генома древней лошади[25]. Возраст образца оценивается в 560—780 тысяч лет. По состоянию на конец 2013 года это самый древний полный ядерный геном. Также были секвенированы ДНК лошади из позднего плейстоцена (~43 тысячи лет назад), пяти современных пород лошадей, лошади Пржевальского и осла. Филогенетический анализ показал, что последний общий предок всего рода Лошади жил 4-4,5 миллиона лет назад, что оказалось вдвое больше принятой до того оценки; популяции предков лошади Пржевальского и домашней лошади разошлись 38-72 тысячи лет назад. В том же году была восстановлена последовательность митохондриальной ДНК пещерного медведя из испанской Пещеры Костей, жившего в среднем плейстоцене, 179—680 тыс. лет назад, причём техника подготовки древней ДНК к секвенированию была оптимизирована для лучшего прочтения коротких (30-50 пн) фрагментов[11]. По состоянию на октябрь 2013-го, древний геном отсеквенирован с покрытием больше единицы только для таких позвоночных, как человек, белый медведь и лошадь. ДНК древних людей Неандертальцы Первым шагом в изучении генетического материала неандертальцев была работа с митохондриальной ДНК, выделенной из костей, которые были обнаружены в 1856 году в долине реки Неандерталь (Германия). В 2006 году был начат проект по секвенированию полного генома неандертальца. В работе использовалась ДНК останков из пещеры Виндия в Хорватии, а также из некоторых других костей. Выравнивание полученного генома вместе с геномом современного человека и геномом шимпанзе, позволило грубо оценить время расхождения современного человека и неандертальца в 270—440 тыс. лет в предположении, что человек и шимпанзе разошлись 6,5 млн лет назад[26]. Геномы неандертальцев из пещеры Сидрон (англ.) (Испания), пещеры Фельдхофер (англ.) (Германия), Мезмайской пещеры (Россия) незначительно отличаются от первого. Секвенирование ДНК древних людей даёт надежду установить функциональные геномные мутации, которые отличают древнего человека от обезьяны, а также проследить отличия современного человека от древних людей. Исследование выявило удивительно малое количество зафиксировавшихся замен в геноме за довольно долгое время. Было найдено всего лишь 5 генов, в которых у современного человека зафиксировалось больше одной замены, меняющей структуру белка (по сравнению с неандертальцем, который в этих локусах совпал с шимпанзе): RPTN (англ.), SPAG17, CAN15, TTF1 (англ.) и PCD16. Сравнение разнообразия SNP для неандертальцев и современного человека позволяет локализовать мишени положительного отбора в геноме. Самый обширный такой участок генома, выявленный анализом различий SNP, принадлежит гену THADA (англ.), и SNP в ближайшем его окружении ассоциированы с диабетом 2 типа, а экспрессия этого гена существенно различается для больных диабетом и здоровых людей. В этом же регионе была обнаружена вставка 9 нуклеотидов в геноме человека, которая отсутствует у всех известных геномов от мыши до приматов и у неандертальца. Также SNP в других регионах ассоциируются с шизофренией, синдромом Дауна, аутизмом. Интерес представляет ген RUNX2 (англ.), который является единственным известным на данный момент геном, связанным с развитием ключично-черепного дизостоза. Анализ SNP также выявляет примесь генетического материала неандертальцев в ДНК неафриканцев, что подтверждает теорию о скрещивании неандертальцев с современными людьми, произошедшем после выхода популяции последних из Африки. Денисовцы Именно анализ древней ДНК позволил обнаружить этот вид древних людей — изначально считалось, что найденные в Денисовой пещере фрагменты скелета (две фаланги пальцев и три коренных зуба) принадлежат неандертальцам. Секвенирование митохондриальной ДНК останков опровергло теорию и показало, что они принадлежат отдельной группе. Различие денисовцев и современных людей в 2 раза превышает различие неандертальцев и современных людей, однако для ядерной ДНК эти различия имеют одинаковый порядок. Возможное объяснение состоит в том, что денисовцы и неандертальцы происходят от общего предка, который ранее отделился от ветви будущего современного человека. Эту гипотезу подтвердило сравнение двух геномов древних людей (денисовец, неандерталец) и пяти геномов современных людей (представителей Франции, Китая, Папуа — Новой Гвинеи, африканских народностей йоруба и бушменов), а также выравнивание геномов денисовцев, неандертальцев и африканцев йоруба на геном шимпанзе. Помимо этого, исследование показало, что денисовцы и неандертальцы являются сестринскими группами. Также было показано, что денисовцы, как и неандертальцы, скрещивались с некоторыми неафриканскими популяциями современных людей: примесь ДНК неандертальцев присутствует во всех неафриканских группах, а примесь денисовцев — у папуасов и меланезийцев[14]. Филогенетические выводы по данным ядерной и митохондриальной ДНК расходятся. Это может объясняться, тем, что мтДНК денисовцев происходит из некой древней линии, которая не прижилась в неандертальцах и современных людях. Большой размер древних популяций делает эту гипотезу правдоподобной, хотя для однозначного разрешения этой проблемы данных пока недостаточно. Гейдельбергский человек Анализ практически полного митохондриального генома гейдельбергского человека из Пещеры Костей позволил оценить возраст находки в 150—640 тысяч лет и показал, что митохондриальные геномы более схожи для гейдельбергского человека и денисовцев, чем для указанных видов и неандертальца, хотя по морфологическим признакам люди из Пещеры Костей близки к неандертальцам. Есть несколько возможных объяснений. Люди из Пещеры Костей могут представлять группу, отличную и от неандертальцев, и от денисовцев, которая внесла вклад в денисовскую мтДНК, но эта версия не объясняет морфологических черт неандертальцев у вида, напрямую с ними не связанного. Второе возможное объяснение состоит в том, что гейдельбергские люди относятся к общим предкам неандертальцев и денисовцев, однако эта версия предполагает существование в этой группе двух сильно разошедшихся линий мтДНК, предковых для неандертальцев и денисовцев. В третьих, не исключено влияние малоисследованных подвидов человека, которые могли сделать вклад в мтДНК гейдельберских людей и денисовцев. Анализ ядерной ДНК может прояснить картину[27]. Древняя эпигенетика Информация о метилировании цитозинов в CpG (англ.) может быть восстановлена непосредственно по данным секвенирования[28]. При дезаминировании, которое происходит случайно с течением времени, неметилированный цитозин превращается в урацил, а метилированный — в тимин. Протокол секвенирования древней ДНК предполагает обработку ДНК урацил-ДНК-гликозилазой и эндонуклеазой VIII, в результате которой урацил удаляется и молекула ДНК раскусывается в соответствующем месте с удалением поврежденного нуклеотида[29]. В результате при секвенировании для тех позиций, где цитозин был метилирован, будет получен большой процент чтений с Т, а неметилированный цитозин прочитается как C по тем молекулам, где дезаминирование в этом месте не произошло. По последовательности ДНК, извлечённой из волос 4000-летних останков палеоэскимоса удалось построить полногеномную карту расположения нуклеосом и метилирования[30], причём профиль метилирования оказался близок к тому, что наблюдается в волосах современного человека. Также восстановлена карта метилирования для неандертальцев и денисовцев[28][31]. Их метилом оказался похож на метилом современного человека, особенно в районе генов «домашнего хозяйства», однако найдено около 2000 областей со значимыми отличиями в метилировании. В частности, у древних людей найдены заметилированные участки в кластере HOXD, задействованном в регуляции развития конечностей, что может объяснять такие анатомические различия между ними и современными людьми, как менее длинные конечности, крупные кисти, широкие локтевые и коленные суставы[28][31]. Патогены Современные методы экстракции и секвенирования древней ДНК позволяют проводить исследования патогенов, полученных из останков давно умерших от болезни людей. Филогенетический анализ полученных образцов позволяет восстанавливать эволюцию болезнетворных организмов. Секвенированы средневековые штаммы чумной палочки и палочки Хансена, а также штамм палочки Коха XIX-го века. Чумная палочка Исследования древней ДНК чумной палочки (Yersinia pestis), полученных из лондонских захоронений жертв «Чёрной смерти» установили, что чумная палочка того времени, если не считать возможных геномных перестроек, характерных для этого вида, и ограничиться однонуклеотидными полиморфизмами, мало отличалась от современных штаммов. Более того, для всех полиморфизмов, отличающих её от современного штамма, в ней присутствовал вариант предка чумной палочки — Y. pseudotuberculosis. Эти данные позволяют предполагать, что генотип древней чумной палочки не являлся главной причиной её высокой вирулентности в то время и, возможно, его вклад был наравне с вкладом таких факторов, как генетически обусловленная восприимчивость носителей, климат, социальные условия, взаимодействия с другими болезнями. При помощи филогенетического анализа был установлен временной интервал, в который жил последний общий предок всех современных патогенных для человека штаммов чумной палочки: 1282—1343 года, причём древняя бактерия оказалась ближе всего к предковому узлу филогенетического дерева[32]. Фитофтора В 2013 году было опубликовано показательное исследование в области секвенирования старых патогенов, посвященное не существующим ныне штаммам фитофторы (Phytophthora infestans), которые вызвали Ирландский картофельный голод в середине XIX века. Генетический материал этих патогенов был выделен из сохраненных листьев картофеля и томата разного времени. До настоящего времени, ввиду отсутствия методов секвенирования, анализ данных был невозможен. Ученые провели анализ разных штаммов патогенов, выделив родственные штаммы из растений в Ирландии, Северной Америки и Европы. Исследования показали, что группа штаммов вызвавшая вышеупомянутый голод (HERB-1) и Северо-Американская группа (US-1) имели последнего общего предка предположительно на территории Мексики на рубеже XVIII и XIX веков. Это исследование особенно интересно с точки зрения подходов к анализу древней ДНК возбудителей прошедших эпидемий как животных, так и растений. Данная область находится на раннем этапе своего развития, однако в случае систематического создания биологических банков, подобные знания могут значительно способствовать ускоренному поиску противомикробных препаратов в ближайшем будущем, в случае возникновения эпидемий, вызванных родственными возбудителями[33]. См. также Примечания - ↑ 1 2 3 4 B. Shapiro, M. Hofreiter (24 January 2014). «A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA». Science 343 no. 6169: 3—16. DOI:10.1126/science.1236573.
- ↑ Higuchi R. et al. DNA sequences from the quagga, an extinct member of the horse family (англ.) // Nature. — 1985. — Vol. 312, no. 5991. — P. 282—284. — DOI:10.1038/312282a0. — PMID 6504142.
- ↑ Pääbo S. Molecular cloning of Ancient Egyptian mummy DNA (англ.) // Nature. — 1985. — Vol. 314, no. 6062. — P. 644—645. — DOI:10.1038/314644a0. — PMID 3990798.
- ↑ Pääbo S. Molecular genetic investigations of ancient human remains (англ.) // Cold Spring Harb Symp Quant Biol. — 1986. — Vol. 51, no. Pt 1. — P. 441—446. — PMID 3107879.
- ↑ 1 2 3 4 5 6 7 8 9 Eske Willerslev and Alan Cooper (January 2005). «Review Paper. Ancient DNA». Proc. R. Soc. B 272 no. 1558: 3—16. DOI:10.1098/rspb.2004.2813.
- ↑ Ermanno Rizzi, Martina Lari, Elena Gigli, Gianluca De Bellis, David Caramelli Ancient DNA studies: new perspectives on old samples (En) // Genetics Selection Evolution. — 2012/12. — Т. 44, вып. 1. — С. 21. — ISSN 1297-9686. — DOI:10.1186/1297-9686-44-21.
- ↑ Pontus Skoglund, Jan Storå, Anders Götherström, Mattias Jakobsson Accurate sex identification of ancient human remains using DNA shotgun sequencing // Journal of Archaeological Science. — Т. 40, вып. 12. — С. 4477–4482. — DOI:10.1016/j.jas.2013.07.004.
- ↑ Adrian M. Whatmore Ancient-Pathogen Genomics: Coming of Age? (англ.) // mBio. — 2014-10-31. — Vol. 5, iss. 5. — P. e01676–14. — ISSN 2150-7511. — DOI:10.1128/mbio.01676-14.
- ↑ Zoe Patterson Ross, Jennifer Klunk, Gino Fornaciari, Valentina Giuffra, Sebastian Duchêne The paradox of HBV evolution as revealed from a 16th century mummy (англ.) // PLOS Pathogens. — 2018-01-04. — Vol. 14, iss. 1. — P. e1006750. — ISSN 1553-7374. — DOI:10.1371/journal.ppat.1006750.
- ↑ 1 2 3 4 Michael Hofreiter, David Serre, Hendrik N. Poinar, Melanie Kuch and Svante Pääbo (2001). «Ancient DNA». Nature Reviews Genetics 2: 353—359. DOI:10.1038/35072071.
- ↑ 1 2 Jesse Dabney et al. (06.08.2013). «Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments». PNAS (201314445): 15758—15763. DOI:10.1073/pnas.1314445110. ISSN 0027-8424.
- ↑ Pontus Skoglund, Bernd H. Northoff, Michael V. Shunkov, Anatoli P. Derevianko, Svante Pääbo, Johannes Krause, and Mattias Jakobsson. (11 February 2014). «Separating endogenous ancient DNA from modern day contamination in a Siberian Neandertal». Proc. Natl. Acad. Sci. USA vol. 111 no. 6: 2229—2234. DOI:10.1073/pnas.1318934111.
- ↑ Этот метод требует наличия референсного генома. В первом приближении метод выглядит следующим образом. Чтения выравниваются на геном, после чего рассматриваются позиции, где встречаются, например, цитозин и тимин. Сравниваются две модели: одна предполагает, что замена цитозина на тимин произошла в результате посмертной деградации ДНК, вторая предполагает, что это полиморфизм или ошибка секвенирования. Модель выбирается по методу максимального правдоподобия.
- ↑ 1 2 David Reich et al. (23/30 December 2010). «Genetic history of an archaic hominin group from Denisova Cave in Siberia». Nature vol. 468: 1053—1060. DOI:10.1038/nature09710.
- ↑ Henrik B. Hansen, Peter B. Damgaard, Ashot Margaryan, Jesper Stenderup, Niels Lynnerup Comparing Ancient DNA Preservation in Petrous Bone and Tooth Cementum (англ.) // PLOS ONE. — 2017-01-27. — Vol. 12, iss. 1. — P. e0170940. — ISSN 1932-6203. — DOI:10.1371/journal.pone.0170940.
- ↑ Jesse Dabney, Michael Knapp, Isabelle Glocke, Marie-Theres Gansauge, Antje Weihmann Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments (англ.) // Proceedings of the National Academy of Sciences. — 2013-09-24. — Vol. 110, iss. 39. — P. 15758–15763. — ISSN 1091-6490 0027-8424, 1091-6490. — DOI:10.1073/pnas.1314445110.
- ↑ Iosif Lazaridis, Dani Nadel, Gary Rollefson, Deborah C. Merrett, Nadin Rohland Genomic insights into the origin of farming in the ancient Near East (En) // Nature. — 2016/08. — Т. 536, вып. 7617. — С. 419–424. — ISSN 1476-4687. — DOI:10.1038/nature19310.
- ↑ Beth Shapiro and Michael Hofreiter (eds.). Ancient DNA: Methods and Protocols. — Methods in Molecular Biology. — © Springer Science+Business Media, LLC 2012. — С. vol. 840. — ISBN DOI 10.1007/978-1-61779-516-9_10.
- ↑ Lindahl T. Instability and decay of the primary structure of DNA (англ.) // Nature. — 1993. — Vol. 362, no. 6422. — P. 709—715. — DOI:10.1038/362709a0. — PMID 8469282.
- ↑ S. Blair Hedges, Mary H. Schweitzer (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1191—1192. DOI:10.1126/science.7761839.
- ↑ В то время сборки человеческого генома ещё не существовало. См. Проект «Геном человека»
- ↑ Zischler H., Höss M., Handt O., von Haeseler A., van der Kuyl A.C., Goudsmit J. (26 May 1995). «Detecting dinosaur DNA». Science 268 no. 5214: 1193—4. DOI:10.1126/science.7605504.
- ↑ Web Miller et al. (20 november 2008). «Sequencing the nuclear genome of the extinct woolly mammoth». Nature vol. 456: 387—392. DOI:10.1038/nature07446.
- ↑ Hendrik N. Poinar et al. (20 January 2006). «Metagenomics to Paleogenomics: Large-Scale Sequencing of Mammoth DNA». Science vol. 311: 392—394. DOI:10.1126/science.1123360.
- ↑ Ludovic Orlando et al. (26 June 2013). «Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse». Nature vol. 499: 74—78. DOI:10.1038/nature12323.
- ↑ Richard E. Green et al. (7 May 2010). «A Draft Sequence of the Neandertal Genome». Science vol. 328: 710—722. DOI:10.1126/science.1188021.
- ↑ Matthias Meyer et al. (16 January 2014). «A mitochondrial genome sequence of a hominin from Sima de los Huesos». Nature 505: 403—406. DOI:10.1038/nature12788.
- ↑ 1 2 3 Elizabeth Pennisi (18 April 2014). «Ancient DNA Holds Clues to Gene Activity in Extinct Humans». Science Vol. 344 no. 6181: pp. 245-246. DOI:10.1126/science.344.6181.245.
- ↑ Briggs et al. (22 Dec 2009). «Removal of deaminated cytosines and detection of in vivo methylation in ancient DNA». Nucl. Acids Res. Vol. 38 (6): pp. e87. DOI:10.1093/nar/gkp1163.
- ↑ Jakob Skou Pedersen et al. (3 December 2013). «Genome-wide nucleosome map and cytosine methylation levels of an ancient human genome». Genome Res 24: 454—466. DOI:10.1101/gr.163592.113.
- ↑ 1 2 David Gokhman, Eitan Lavi, Kay Prüfer, Mario F. Fraga, José A. Riancho, Janet Kelso, Svante Pääbo, Eran Meshorer, Liran Carmel (April 17 2014). «Reconstructing the DNA Methylation Maps of the Neandertal and the Denisovan». Science Published Online. DOI:10.1126/science.1250368.
- ↑ Kirsten I. Bos, Verena J. Schuenemann, G. Brian Golding, Hernán A. Burbano, Nicholas Waglechner A draft genome of Yersinia pestis from victims of the Black Death (En) // Nature. — 2011/10. — Т. 478, вып. 7370. — С. 506–510. — ISSN 1476-4687. — DOI:10.1038/nature10549.
- ↑ Kentaro Yoshida Verena J Schuenemann Liliana M Cano Marina Pais Bagdevi Mishra Rahul Sharma Chirsta Lanz Frank N Martin Sophien Kamoun Johannes Krause Marco Thines Detlef Weigel Hernán A Burbano The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine (англ.) // eLife : Published online. — 2013. — Май.
Литература wikiredia.ru
|
